1.本发明属于法医学领域,具体涉及利用焦磷酸测序和随机森林回归分析进行年龄预测的方法。
背景技术:
2.对未知样本捐赠者的生理年龄评估是法医调查中最重要的工具之一。它缩小了犯罪嫌疑人的范围,进而对罪犯的外部可见特征预测和生物地理祖先推断进行补充。先前建立的年龄分类方法涉及对骨骼特征的形态学分析。当骨骼和牙齿等固体组织可用时,可通过人类学方法精确地确定年龄。然而,由于在法医调查过程中更容易遇到其他组织,如体液,因此在实践中很难使用此类方法。最近,提出了几种基于分子水平的方法来估算年龄,包括端粒长度分析,线粒体dna的年龄依赖性缺失或t细胞dna重排,以及蛋白质改变,如天冬氨酸的外消旋作用和晚期糖基化终产物。然而,所有这些方法都有局限性,限制了它们在犯罪现场的适用性,特别是它们的低准确性和严格的样本要求。例如,基于信号联合t细胞受体重排切除环(sjtrecs)量化的年龄预测标准误差为
±
8.0年。
3.这些方法的一个可能替代方法是检测表观遗传修饰(例如甲基化),现在已知这些修饰可随年龄变化。迄今为止,法医学年龄预测的研究主要集中在全血样本上,平均绝对偏差(mad)为3-10年,主要采用多元线性回归模型。少量研究使用机器学习算法,如支持向量机(svm)、人工神经网络(ann)和随机森林回归(rfr),实现了相对较低的预测误差(3.24-4.7年);然而,这些研究仅在新鲜体液中进行。此外,基于斑痕的年龄预测(在犯罪现场调查中更常见)尚未得到系统研究。
4.因此,本发明旨在建立一种灵敏、快速、可靠的基于焦磷酸测序技术和随机森林回归计算模型,适用于包括血痕在内的各种检材的年龄预测方法。
技术实现要素:
5.本发明在基因组序列中筛选出一套用于分析法医学案件中检材的dna甲基化年龄预测位点,并对每一位点设计了引物,使用焦磷酸测序技术对各位点甲基化水平进行分析,而后利用随机森林回归分别为男性和女性建立年龄预测模型。旨在发明一种灵敏、快速、可靠且使用较少位点仍能保持高准确度的年龄预测分析方法,该检测方法可用于血痕等检材的年龄预测,在该方法中我们对dna提取、引物设计和测序方案都进行了优化。
6.术语:
7.rfr:random forest regressor,随机森林回归。
8.svr:support vector regression,支持向量回归。
9.mad:mean absolute deviation,平均绝对误差。
10.一方面,本发明提供了一种用于年龄预测的方法。
11.所述的方法中包括焦磷酸测序和随机森林回归分析,所述的随机森林回归分析模型使用r package random forest构建,并采用正向选择法确定最佳的位点组合。
12.所述的方法中随机森林回归分析模型构建中的参数设置:mtry参数与每次建模的cpg位点数相同,最小节点大小为5,树的数量设置为1000。
13.所述的随机森林回归分析模型选用与年龄相关的dna甲基化标记分别位于elovl2、c1orf132、trim59、klf14、fhl2和nptx2基因。
14.所述的随机森林回归建立年龄预测模型时,共选用7个与年龄相关的dna甲基化位点,其中男性3个为:trim59.pos7、klf14.pos2、elovl2.pos7;女性4个为trim59.pos8、klf14.pos3、clorf132.pos2和fhl2.pos6。
15.所述的焦磷酸测序中使用的pcr产物的体积为12μl。
16.在一些实施例中,所述的方法中包括以下步骤:
17.(1)dna提取;
18.(2)亚硫酸盐转化;
19.(3)pcr;
20.(4)焦磷酸测序;
21.(5)模型预测。
22.另一方面,本发明提供了一组用于随机森林回归分析进行年龄预测的基因组合。
23.所述的基因组合中包括elovl2、c1orf132、trim59、klf14、fhl2和nptx2。
24.所述的甲基化位点中包括男性相关位点:trim59.pos7、klf14.pos2、elovl2.pos7和女性相关位点:trim59.pos8、klf14.pos3、clorf132.pos2、fhl2.pos6。
25.再一方面,本发明提供了一组用于随机森林回归分析进行年龄预测的引物。
26.所述的引物用于焦磷酸测序。
27.所述的引物及其测序位点如下:
[0028][0029]
其中,引物序列f、r、s分别代表正向引物、反向引物和测序引物,序列前标记biotin表示引物带有生物素标记。
[0030]
又一方面,本发明提供了前述的方法和/或基因组合和/或甲基化位点和/或引物在制备用于预测年龄的试剂盒中的应用。
[0031]
又一方面,本发明提供了一种用于预测年龄的试剂盒。
[0032]
所述的试剂盒中包括以下引物:
[0033]
[0034][0035]
所述的试剂盒中还包括其他用于焦磷酸测序的试剂。
[0036]
所述的试剂盒与随机森林回归模型联合使用。
[0037]
本发明的有益效果:
[0038]
(1)仅需0.1ng模板dna,可用于难度较高的法医血痕检材
[0039]
许多技术,如epityper、snapshots、焦磷酸测序和大规模平行测序(mps)都可以提供较准确的dna甲基化测量方法。而限制epityper分析法在法医学中应用的一个主要原因是其需要高达1μg的基因组dna,然而实际犯罪现场调查中很难获得如此高量的dna,往往在犯罪现场更常遇见体液斑迹。相较于epityper,mps所需的模板dna可降至10ng,snapshots需要4ng模板dna。而在本发明中,使用0.1ng的模板dna即可进行准确的年龄预测。先前的研究表明基于甲基化进行成功的年龄预测需要10-20ng模板dna。因此,本发明的检测方法在现有的血痕检测中具有最高的灵敏度,并具有良好的法医学应用前景。
[0040]
(2)整个过程可在10小时内完成
[0041]
本发明的方法可在一天内完成,远远快于其他可用的方法。dna提取/定量、硫酸氢钠转化、pcr和焦磷酸测序试验分别需要2h、2.5h、3h和2h。相比之下,epityper和mps的标准程序都需要2天以上的时间。特别是,mps需要专门的设备和复杂的生物信息学分析系统,难以在3天内完成。
[0042]
(3)针对性别差异,分性别建立两个独立的年龄预测模型
[0043]
选择随机森林回归(random forest regression,rfr)建立年龄预测模型,分别使用男性3个(trim59.pos7、klf14.pos2、elovl2.pos7)和女性4个(trim59.pos8、klf14.pos3、clorf132.pos2和fhl2.pos6)位点,共7个位点的最终模型为男性和女性的预测平均绝对误差(mad)分别为2.8年(r=0.99)和2.93年(r=0.98)。
[0044]
(4)仅使用3-4个cpg位点,预测年龄的准确性可达到mad<3年
[0045]
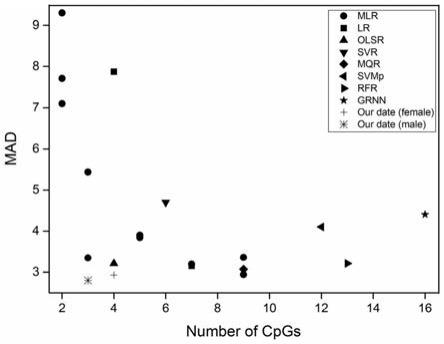
对过去几年的年龄预测研究进行的荟萃分析表明,先前研究建立的年龄预测模型,几乎所有mad的年龄均》3年。由于使用rfr,我们的模型是最有效的(mad《3年,且只需3-4个cpg位点,男性样本仅使用3个cpg位点,女性样本仅使用4个cpg位点)。本发明的位点少且仍能保持高准确度的年龄预测模型对于法医推断更为实用。
附图说明
[0046]
图1为随机森林回归(rfr)在年龄预测方面优于支持向量回归(svr)。
[0047]
图2为随机森林回归(rfr)测试数据集的预测年龄与实际年龄。
[0048]
图3为微量dna中7种甲基化标记的灵敏度检测。
[0049]
图4为7个cpg位点甲基化水平与年龄的相关性分析。
[0050]
图5为与已发表研究的年龄预测方法准确度比较。
具体实施方式
[0051]
下面结合具体实施例,对本发明作进一步详细的阐述,下述实施例不用于限制本发明,仅用于说明本发明。以下实施例中所使用的实验方法如无特殊说明,实施例中未注明具体条件的实验方法,通常按照常规条件,下述实施例中所使用的材料、试剂等,如无特殊说明,均可从商业途径得到。
[0052]
实施例1dna提取及位点筛选
[0053]
(1)dna提取:
[0054]
优化dna提取方案,减少血痕微量dna损失。
[0055]
甲基化分析的准确性取决于从血迹中提取高质量的dna。qiaamp dna investigator kit已被认为是从法医样本中提取dna的更可靠方法,可在2小时内获得成功提取出高质量的dna。我们对该试剂盒进行了进一步优化,包括在较高温度下缩短孵育时间、在裂解液中添加载体rna以及加热溶解dna的试剂。
[0056]
先前的方法是样本在56℃下孵育1小时,改进后的方法是样本在85℃下孵育10分钟,然后在56℃下二次孵育1小时,以此增加血痕dna的提取量。
[0057]
加热溶解dna的试剂也可以加速血斑上的细胞脱落和增加dna溶解,以此来减少微量dna的损失。
[0058]
(2)位点选择:
[0059]
根据文献,选择了六个与年龄相关的dna甲基化标记,分别位于elovl2、c1orf132、trim59、klf14、fhl2和nptx2,以确保我们关注的是与年龄相关的区域。
[0060]
(3)引物设计:
[0061]
由于从血痕中获得的dna数量极少且质量较低,pcr的准确性和敏感性至关重要。使用pyromark assay design version 2.0(qiagen,德国)设计pcr引物和测序引物。设计引物时,对目标序列进行调整,使引物包含尽可能多的胞嘧啶(c),以检测更多的甲基化位点。我们避免了目标区域的snp和其他多态性,因为它们可能会导致测序反应出现偏差。此外,排除引物结合序列中可能的甲基化位点,将gc含量保持在60%以下,选择具有高特异性的引物(即不形成引物二聚体)。必要时,我们改变已公布的方法(例如,添加二甲基亚砜(dmso)以避免二聚体形成)以优化方案。pcr的引物中,一条引物的5’端需使用生物素标记,以与链霉亲和素包被的磁珠结合,用于后续单链pcr产物的分离纯化,另一条不要标记。生物素标记的引物中含有游离的生物素,游离生物素会与模板竞争结合到链霉亲和素包被的磁珠上,而降低信号水平,须使用hplc纯化的生物素标记的引物。每个目的基因的扩增子长度范围为105-306bp。最终得到的引物如下表所示:
[0062]
表1年龄相关甲基化分析的pcr引物、焦磷酸测序引物和cpg序列
[0063][0064][0065]
实施例2焦磷酸测序技术检测dna甲基化
[0066]
(1)亚硫酸氢盐转化
[0067]
使用epitect fast dna亚硫酸氢盐试剂盒(德国,qiagen)对提取的dna(40μl)进行亚硫酸氢盐转化。将dna样本与ct转化试剂(亚硫酸氢盐试剂盒)混合以获得最终体积为140μl的产物,然后在95℃下孵育5分钟,60℃20分钟,然后纯化。
[0068]
(2)pcr
[0069]
反应混合物(25μl)包含2μl转化dna、12.5μl pcr预混物(德国,qiagen)和0.1-0.5mm引物。调整引物浓度以获得不含二聚体的特异性dna产物。热循环条件如下:95℃变性10分钟;在95℃下进行45次循环,持续30秒,在56℃下进行30秒(nptx2 58℃,30秒),在72℃下进行30秒;然后在72℃下进行5分钟的最终延伸。使用琼脂糖凝胶电泳进行电泳检测。
[0070]
(3)焦磷酸测序
[0071]
使用pyromark q48热测序仪(德国,qiagen)和pyro-gold试剂盒(德国,qiagen)对生物素标记的pcr扩增产物制备的模板进行测序。先前的焦磷酸测序过程中,pcr产物的体积为10μl,会产生无法与背景信号明确区分的不稳定信号。我们的方法将pcr产物的体积增加到12μl,可有效避免不稳定信号的产生。
[0072]
实施例3构建血痕年龄预测模型
[0073]
(1)对比svr和rfr模型的年龄预测准确性
[0074]
我们先前的研究结果表明,svr模型比多元线性回归、多元非线性回归和反向传播
神经网络等方法更精确,因此,我们利用svr和rfr模型,基于所有46个cpg位点进行组合,建立最佳拟合年龄预测模型,并计算其预测精度。svr模型是在r package e1071中构建,参数设置:cost=2,gamma=0.8,epsilon=0.1。rfr模型用r package random forest构建,mtry参数与每次建模的cpg位点数相同,最小节点大小为5,树的数量设置为1000。
[0075]
为了提高计算速度,采用正向选择法确定最佳的位点组合。从241个血痕样本(年龄范围为10-79岁的241名健康中国汉族志愿者,其中包括128名男性和113名女性的全血样本。所有捐助者都提供了知情同意书,中国科学院北京基因组研究所通过了这项研究的伦理批准)中随机抽取70%的样本形成训练数据集,剩余的30%作为测试数据集,以评估rfr模型的准确性。训练重复100次,每次选择最佳位点(即最小mad)。选择记录频率最高的位点作为最终模型的合适位点。在双位点训练模型中,在最佳位点之后,记录频率最多、mad最小的位点作为第二最佳位点。
[0076]
rfr构建的年龄预测模型,女性使用4个位点(trim59.pos8、klf14.pos3、clorf132.pos2和fhl2.pos6)男性使用3个位点(trim59.pos7、klf14.pos2、elovl2.pos7),所得的mads《3年。在svr模型下,即使男性和女性都有8个位点,mad稳定在4.5年左右,这一结果表明,rfr在年龄预测方面优于svr(图1)。
[0077]
(2)测试数据集验证预测准确性
[0078]
剩余的30%的血痕样本(男性38名,女性33名)作为测试数据集,在rfr模型中验证最终模型筛选出的7个位点(男性3个位点:trim59.pos7、klf14.pos2、elovl2.pos7;女性4个位点:trim59.pos8、klf14.pos3、clorf132.pos2和fhl2.pos6)的年龄预测准确性,得出男性和女性的预测mad分别为2.8年(r=0.99)和2.93年(r=0.98)(图2)。
[0079]
实施例4灵敏度检测
[0080]
收集年龄范围为10-79岁的241名健康中国汉族志愿者(128名男性和113名女性)的全血样本。所有捐助者都提供了知情同意书,中国科学院北京基因组研究所通过了这项研究的伦理批准。
[0081]
将20μl全血等分到滤纸上制备血迹,然后在室温下保存1年。为了确定检测灵敏度,将从血痕中提取的dna连续稀释至100、50、10、5、2.5、1.0、0.50、0.25和0.10ng。不同浓度的血痕样本均进行甲基化分析,先进行亚硫酸氢盐转化,然后进行pcr扩增和焦磷酸测序(参照实施例1的方法)。对比0.1ng dna和较高dna浓度之间甲基化百分比的差异,判定我们所提出的甲基化检测方法在血痕检测中的灵敏度。
[0082]
我们观察到用于年龄预测的女性4个cpg位点(trim59.pos8、klf14.pos3、clorf132.pos2和fhl2.pos6)和男性3个cpg位点(trim59.pos7、klf14.pos2、elovl2.pos7),0.1ng dna与较高浓度dna之间的甲基化百分比无显著差异(p≥0.05,ks检验;图3)。elovl2.pos7位点,需要1.0ng dna能达到相似的水平。
[0083]
实施例5dna甲基化水平与年龄的相关性分析
[0084]
采集年龄范围为10-79岁的241名健康中国汉族志愿者(128名男性和113名女性)的全血样本,制备成血痕样本并进行甲基化分析,将本发明中位点与年龄进行相关性分析。本发明最终形成的血痕年龄预测模型包含跨越3个基因的7个cpg位点,3个已知位点,4个新cpg位点。结果表明,其中的5个cpg位点来自3个基因(trim59、klf14和c1orf132),在中国受试者的血痕分析中与年龄相关(图4)。
[0085]
实施例6对比已发表研究的年龄预测准确度
[0086]
对过去多年的年龄预测研究进行的荟萃分析表明,几乎所有mad的年龄均》3年(图5)。与之前的研究相比,由于使用rfr,我们的模型是最有效的(mad《3年,只需3-4个cpg位点)。图5中,实心点代表已公布的结果,不同模型中的数学方法以不同的形状表示。而“十”字和“米”字符号分别代表我们对女性和男性建立的年龄预测结果。
