一种用于香菇高多糖菌株早期筛选的indel标记组合及其检测方法
技术领域
1.本发明涉及分子遗传育种技术领域,具体的说涉及一种用于香菇高多糖菌株早期筛选的indel标记组合及其检测方法。
背景技术:
2.香菇已成为我国生产区域最广、总产最高、影响力最大的主要栽培菇类,体现出显著的社会效益,随着“健康中国”国家战略的实施及“药食同源、健康养生”理念日益深入人心,多糖类产品成为香菇生产和消费新的生长点,也为延长香菇产业链、消解香菇产能、提升产业能级提供了新机会。香菇多糖的开发与利用效益受原料香菇的多糖含量及品质的影响极大。新品种、新技术的开发对香菇生物活性物质的产业化与实际应用,乃至整个香菇产业的发展,具有至关重要的战略意义。生物活性多糖是一种次生代谢物质,它们在生物体中的含量一般都较低,大规模产业化开发成本均较高,这是当前国内外在多糖类物质产业化研发过程中遇到的主要瓶颈问题之一。高多糖香菇新品种的选育成为解决这一瓶颈问题的根本途径。
3.目前的香菇育种工作中,对亲本的选择和选育后代的筛选一直是难点,存在很大的盲目性。对大量的选育后代进行筛选时,需要大量的小试重复,非常耗时费力。通过分子标记辅助进行定向选择具有特定性状的亲本以及选育后代的方法,在作物育种中已有大量的应用,然而这种方法在食用菌中还没有使用。随着分子标记技术的成熟及香菇基因组测序的完成,利用分子标记进行定向选育在香菇等食用菌中的应用已是必然。indel分子标记是一种基于高通量测序技术的应用,具有数量丰富、准确性高、稳定性好、分型快捷简便等优点。
4.因此,开发一种用于香菇高多糖菌株早期筛选的indel标记组合成为本领域的主要研究问题。
技术实现要素:
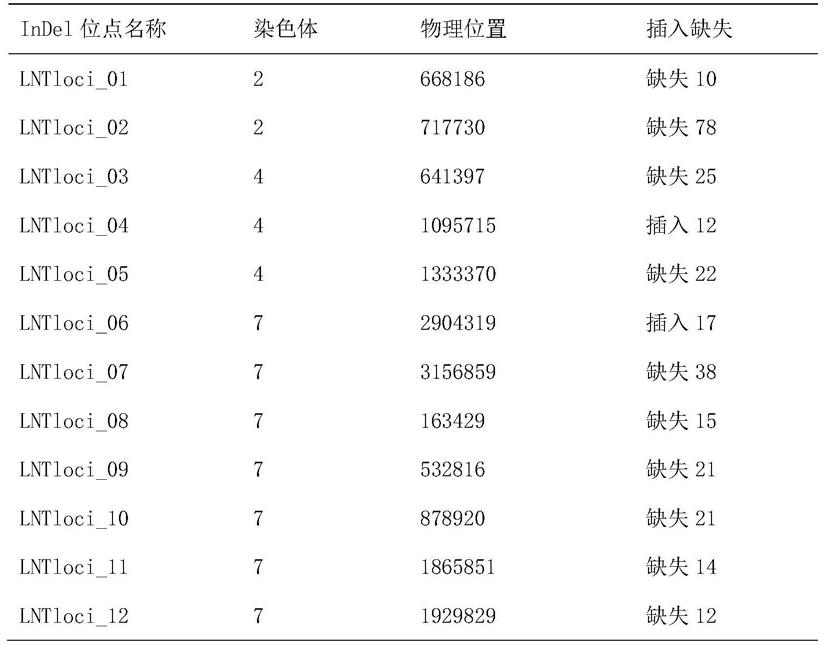
5.本发明的目的之一在于提供一种用于香菇高多糖菌株早期筛选的indel标记组合,所述indel标记组合包含12个indel标记,编号分别为lntloci_01~lntloci_12,所述的indel位点所在的染色体、具体物理位置以及插入缺失信息如表1所示,
6.表1香菇高多糖菌株indel标记组合
[0007][0008]
本发明还提供了上述用于香菇高多糖菌株早期筛选的indel标记组合的检测方法,所述方法包括以下步骤:
[0009]
(4)提取所要检测菌株的dna;
[0010]
(5)对所要检测的香菇菌株进行pcr扩增;
[0011]
(6)确定检测菌株在表1中12个indel位点的基因型;
[0012]
其中,步骤(2)中进行pcr扩增需要的特异性引物为seqno.1~24的引物对,具体的引物信息如表2所示,
[0013]
表2 12个indel位点的基因型的等位基因特异性引物
[0014]
[0015][0016]
本发明还提供了一种香菇菌株多糖含量高低的检测方法(或者一种香菇高多糖菌株的筛选方法),该方法包括如下步骤:
[0017]
(3)检测待检菌株的lntloci_01~lntloci_12基因分型;
[0018]
(4)与indel标记组合基因分型hhabababaaah进行比较,待测菌株与高多糖标准基因分型或者pcr扩增条带拥有75%以上的相似度即为高多糖含量菌株,25%以下的相似度即为低多糖含量菌株。
[0019]
其中,indel标记组合基因分型hhabababaaah(h代表可扩增条带(1+2),a代表可扩增条带1,b代表可扩增条带2)是香菇菌株的高多糖标准基因分型,该高多糖标准基因分型的pcr扩增条带编号组合为:(1+2)(1+2)121212111(1+2)。
[0020]
本发明还提供了上述用于香菇高多糖菌株早期筛选的indel标记组合的应用,其可用于香菇分子标记辅助育种,或可用于香菇高多糖菌株鉴定及选育子代高多糖菌株筛选。
[0021]
本发明以香菇育种群体为研究对象,综合利用混池重测序和生物信息学手段,对高多糖优异标记位点进行开发,形成12对高多糖菌株早期筛选的indel标记组合和12个indel位点的基因型的等位基因特异性引物,以及该标记组合在判定香菇子实体多糖含量高低方面的应用方法,利用该标记组合和判定方法能够快速鉴定香菇子实体高多糖育种材料和育种后代,检测准确性高,可重复性好,检测效率高,操作性强,克服了现有技术只能在
子实体成熟期才能进行子实体多糖含量测定的育种效率低下的问题,实现在菌丝阶段就能进行高低多糖菌株鉴定的技术效果。
附图说明
[0022]
图1香菇沪香f2菌株的杂交子代菌株多糖含量分布图
[0023]
图2高多糖标准基因分型的indel标记组合pcr扩增图谱
[0024]
其中m是d2000 bpdna ladder,数字1-12代表的是所用的12对indel标记引物图3实施例3的12个菌株的pcr扩增结果
具体实施方式
[0025]
以下结合具体实施例进一步说明本发明。实施例仅用于说明本发明而不用于限制本发明的范围。此外,应理解在阅读了本发明的内容之后,本领域技术人员可以对本发明做各种改动和修改,这些等价形式同样落于本技术所附权利要求书所限定的范围。
[0026]“沪香f2”菌株:上海市认定品种,保藏于上海市农业科学院食用菌研究所。
[0027]
marker、premix taq
tm
均购自:takala bio宝日生物科技有限公司。
[0028]
核酸染料购自:北京全式金生物技术有限公司。
[0029]
其余材料和试剂均为普通市售产品。
[0030]
实施例1
[0031]
确定本发明的用于香菇高多糖菌株早期筛选的indel标记组合和标记引物
[0032]
取长势良好、菌盖完全打开、菌褶明显的亲本“沪香f2”菌株,构建f2自交群体,共设计了1028个自交配对组合,其中201个自交后代能正常出菇,正常出菇的子实体均在生长至菌膜将破时进行采收,将采收的香菇子实体,去柄留盖,放置干燥箱中50℃烘干至恒重,粉碎过80目筛,最终共收集到191个自交后代子实体粉末。
[0033]
采用苯酚-硫酸法测定自交子代多糖含量。自交子代多糖含量分布如图1所示。
[0034]
根据子代多糖含量结果,分别选取30株极端高多糖含量菌株和30株极端低多糖含量菌株,将菌丝接种至pda培养基中,23-25℃避光培养,10d后收集菌丝,采用ctab法提取香菇总dna。
[0035]
将dna样品送至北京百迈克生物科技有限公司测序。
[0036]
原始测序数据由测序公司进行质量控制,过滤掉接头序列、低质量和长度小于50bp的reads,生成高质量序列。采用bwa(v0.7.12)软件将过滤后的数据比对到香菇参考基因组上;采用gatk(v4.0.3.0)软件进行indel变异检测,对检测indel位点进行过滤,共得到71677个群体indel。取深度大于10的snp位点用于ed(欧氏距离)值的计算,以snp的ed值的平方在染色体上的分布作图,用loess曲线拟合。结果显示:2、4、7号染色体上出现显著峰,表明这三条染色体上存在与多糖含量相关的位点。
[0037]
进一步与参考基因组比对,基于snp位点的缺失及比例的不同,定位到7个与多糖含量相关的位点,分别位于2号染色体的180000~766000bp、5160000~5180000bp,4号染色体的26800-1977000~bp、2810000-3200000bp,7号染色体的40000~940000bp、1840000~2000000bp、2810000~3200000bp~。对上述7个位点的插入、缺失片段进行检测,共筛选到47个与多糖含量相关的indel标记。设计并合成47对引物,对60株菌株进行基因分型。根据
基因分型结果和多糖含量性状筛选出如表1的12个高多糖含量indel标记组合和如表2的12对标记引物。
[0038]
实施例2应用实施例1得到的indel标记组合对香菇菌株多糖含量高低进行检测
[0039]
对实施例1中的30株高多糖菌株和30株低多糖菌株提取的dna采用筛选出的12对标记引物进行pcr扩增,其中pcr反应条件如下:
[0040]
pcr扩增反应体系为总体积20μl,由2
×
pcr premix taq
tm
(1.25u/25μl taq dna酶,0.4mm/l dntp,0.3mm/l pcr buffer)l0μl,l0μmol/l indel标记正向引物和反向引物(序列见表1)各lμl,浓度20-30ng/μl提取的模板dna 2μl,ddh2o 6μl。
[0041]
pcr反应条件:94℃预变性5min,94℃变性1min,55℃退火1min,72℃延伸1min,35个循环;72℃保持5min,10℃保存。
[0042]
将上述pcr扩增得到的产物,点样5μl于核酸染料的琼脂糖凝胶上电泳,琼脂糖凝胶的体积百分比浓度为2.5%,电泳缓冲液为1
×
tae,电压90v下电泳5h,拍照,对条带进行统计分析,确定高多糖菌株的indel标记标准基因分型为:hhabababaaah,其pcr扩增条带如图2所示,条带编号组合为:(1+2)(1+2)121212111(1+2)。
[0043]
191个自交子代的子实体多糖含量分布如图1所示,分布范围在4.0~13.2g/100g,30株低多糖菌株的多糖含量分布范围在3.989~6.971g/100g;30个高多糖菌株的多糖含量分布范围在9.870~13.208g/100g。
[0044]
与高多糖标准基因分型或者pcr扩增条带拥有50%以上的相似度的菌株占30株高多糖含量菌株的70%,有4株高多糖菌株与标准菌株相似度仅为25%;与高多糖标准基因分型或者pcr扩增条带拥有35%以下的相似度的菌株占30株低多糖含量菌株的77%,有1株低多糖菌株与标准菌株相似度达到了75%;所以与高多糖标准基因分型或者pcr扩增条带拥有75%以上的相似度的菌株确定为高多糖含量菌株,而与高多糖标准基因分型或者pcr扩增条带拥有25%以下的相似度的菌株确定为低多糖含量菌株。
[0045]
实施例3
[0046]
随机选取构建的自交群体子代高多糖菌株4株、低多糖菌株8株,将菌丝接种至pda培养基中,23-25℃避光培养,10d后收集菌丝,采用ctab法提取香菇总dna。采用表2的12对标记引物进行pcr扩增,其中pcr反应条件同实施例2。
[0047]
将上述pcr扩增得到的产物,点样5μl于核酸染料的琼脂糖凝胶上电泳,琼脂糖凝胶的体积百分比浓度为2.5%,电泳缓冲液为1
×
tae,电压90v下电泳5h,拍照,pcr扩增图谱如图3所示,对条带进行统计分析。
[0048]
统计结果如表3所示,按照本发明的一种香菇高多糖菌株的筛选方法,即待测菌株与高多糖标准基因分型或者pcr扩增条带拥有75%以上的相似度即为高多糖含量菌株,25%以下相似度即为低多糖菌株,除了1583外,其余菌株判定结果符合这些菌株在该群体中的多糖含量测定结果。
[0049]
表3 12株菌株pcr扩增条带带型统计结果
[0050][0051]
