染色体外dna鉴定和使用方法
1.相关申请
2.本技术根据35 u.s.c.
§
119(e)要求2019年4月30日提交的美国临时申请no.62/840,735的权益,其全部内容通过引用整体并入本文。
3.政府利益
4.本发明在由国立卫生研究院(national institutes of health,nhi)授予的nci p30ca034196、u54 dk107967、um1 hg009409和r01 ca190121基金的政府支持下完成。政府对本发明享有一定的权利。
技术领域
5.在一些方面,本发明涉及鉴定染色体外环状dna(extrachromosomal circular dna,ecdna)的方法和使用ecdna评估靶基因转录的方法。
背景技术:
6.染色体外dna(extrachrosomal dna,ecdna)是染色体外环状染色质元件[cox,d.et al.(1965)lancet 1,55-58;spriggs,a.i.et al.(1962)br med j 2,1431-1435]。最初通过显微成像被描述为在细胞核型中的“双微染色质体(double-minute chromatin body)”[cox,d.et al.(1965)lancet 1,55-58],ecdna被认为是与体外耐药性相关的基因扩增模式[spriggs,a.i.et al.(1962)br med j 2,1431-1435;decarvalho,a.c.etal.(2018)nat genet50,708-717;nathanson,d.a.et al.(2014)science 343,72-76]。最近,发现ecdna在原发性癌症中很常见[xu,k.et al.(2018)acta neuropathol,doi:10.1007/s00401-018-1912-1],并构成了癌基因扩增的真正机制和适应性储库[turner,k.m.et al.(2017)nature 543,122-125]。ecdna可来源于染色体碎裂的(chromothriptic)基因组破碎(genome shattering)事件,并且因此可由数百个dna片段组成[ma,k.et al.(2012)int j mol sci 13,11974-11999]。其在癌细胞中的积累为响应于肿瘤微环境中的选择性压力以及响应于细胞毒性治疗剂提供了竞争优势[spriggs,a.i.et al.(1962)br med j 2,1431-1435;kohl,n.e.et al.(1983)cell 35,359-367]。由不连贯的遗传模式导致的ecdna水平的快速波动[spriggs,a.i.et al.(1962)br med j 2,1431-1435]可能有助于肿瘤进化的机制。胶质母细胞瘤(glioblastoma,gbm)是一种侵袭性脑肿瘤,在其中经常观察到ecdna[rausch,t.et al.(2012)cell 148,59-71;xue,y.et al.(2017)nat med 23,929-937]。先前使用标准基因组方法对一组独特的gbm来源神经球培养物进行的分析检测到多个含有癌基因(包括egfr、myc和cdk4)的ecdna[spriggs,a.i.et al.(1962)br med j 2,1431-1435]。虽然已开始通过这些标准方法对ecdna的存在及其结构信息进行表征,但尚未了解ecdna被用于调节癌症进展以及被用于导致癌症耐药性的机制。
技术实现要素:
[0007]
根据本发明的一个方面,提供了鉴定细胞中染色体外dna(ecdna)的方法,所述方
法包括:(a)检测非线性dna分子与染色体对中至少一个线性染色体之间的染色质相互作用,其中相互作用包括非线性dna分子与染色体对中至少一个线性染色体之间的接触,并且其中存在以下项则将非线性dna分子鉴定为ecdna:(i)在细胞中所检出的染色质相互作用的显著高频率;(ii)非线性dna分子与细胞中每个染色体对中至少一个线性染色体之间的接触;以及(iii)在多个细胞中非线性dna分子的平均每细胞拷贝数随时间的增加。在一些实施方案中,所述方法还包括确定所检出的染色质相互作用的频率。在某些实施方案中,所述方法还包括确定非线性dna的大小。在一些实施方案中,所述方法还包括确定细胞中非线性dna分子的拷贝数。在一些实施方案中,所述方法还包括在第一时间确定多个细胞中非线性dna分子的平均每细胞拷贝数,并将所确定的平均值与非线性dna的对照平均每细胞拷贝数进行比较。在某些实施方案中,非线性dna分子的对照平均每细胞拷贝数是在不同时间点在所述多个细胞中确定的非线性dna分子的平均每细胞拷贝数。在一些实施方案中,所述方法还包括确定非线性dna的至少一部分的序列。在某些实施方案中,所述方法还包括在所确定的序列中鉴定癌基因序列的存在。在一些实施方案中,细胞是癌细胞。在一些实施方案中,细胞是癌前细胞。在一些实施方案中,细胞从包含癌细胞的多个细胞获得。在某些实施方案中,细胞从对象获得。在一些实施方案中,对象是以下中的至少一种:被诊断患有癌症、被怀疑患有癌症和处于患癌症的风险中。在一些实施方案中,细胞从细胞培养物获得。在某些实施方案中,细胞是脊椎动物细胞。在一些实施方案中,细胞是哺乳动物细胞,并且任选地是人细胞。
[0008]
根据本发明的另一方面,提供了鉴定受ecdna调节的癌基因的方法,所述方法包括:(a)检测ecdna与位于细胞中的一种或更多种靶基因之间的相互作用,其中所述检测包括直接测量ecdna与一种或更多种靶基因的调节元件之间的染色质相互作用;(b)鉴定步骤(a)中检出的相互作用的靶基因中的一种或更多种,其中靶基因的转录受所检出的相互作用的调节;以及(c)确定步骤(b)中鉴定的靶基因中的一种或更多种是否为癌基因,其中靶基因被确定为癌基因则将该癌基因鉴定为受ecdna调节的癌基因。在一些实施方案中,步骤(b)中的鉴定包括测量所鉴定的靶基因的转录水平,并将所测得的水平与靶基因的对照转录水平进行比较。在某些实施方案中,转录调节是转录提高。在一些实施方案中,步骤(a)中的检测手段包括chia-pet方法。在一些实施方案中,步骤(a)中的检测手段包括hi-c方法。在一些实施方案中,调节元件包括靶基因的启动子。在某些实施方案中,靶基因位于线性染色体上。在一些实施方案中,靶基因位于ecdna上。在某些实施方案中,靶基因位于第二ecdna上。在一些实施方案中,细胞是癌细胞。在一些实施方案中,细胞是癌前细胞。在某些实施方案中,细胞从包含癌细胞的多个细胞获得。在一些实施方案中,细胞从对象获得。在一些实施方案中,对象是以下中的至少一种:被诊断患有癌症、被怀疑患有癌症和处于患癌症的风险中。在一些实施方案中,细胞从细胞培养物获得。在某些实施方案中,细胞是脊椎动物细胞。在一些实施方案中,细胞是哺乳动物细胞,并且任选地是人细胞。
[0009]
根据本发明的另一方面,提供了确定癌症的癌基因状态的方法,所述方法包括:(a)在癌细胞中鉴定受ecdna调节的癌基因,以及(b)确定癌基因的水平和ecdna调节作用中的一种或更多种,以作为对癌症的癌基因状态的确定。在一些实施方案中,步骤(a)中的鉴定手段包括:(i)检测ecdna与位于癌细胞的dna中的一种或更多种靶基因之间的相互作用,其中所述检测包括直接测量ecdna与一种或更多种靶基因的调节元件之间的染色质相互作
用;(ii)鉴定在步骤(i)中检出的相互作用中的靶基因,其中该靶基因的转录受所检出的相互作用的调节;以及(iii)确定步骤(ii)中鉴定的靶基因中的一种或更多种是否为癌基因,其中靶基因被确定为癌基因则将该癌基因鉴定为癌细胞中受ecdna调节的癌基因。在某些实施方案中,步骤(ii)中的所述鉴定包括测量所鉴定的靶基因的转录水平,并将所测得的水平与靶基因的对照转录水平进行比较。在一些实施方案中,转录调节是转录提高。在一些实施方案中,步骤(i)中的检测手段包括chia-pet方法。在一些实施方案中,步骤(i)中的检测手段包括hi-c方法。在某些实施方案中,调节元件包括靶基因的启动子。在一些实施方案中,启动子的激活提高靶基因的转录。在一些实施方案中,靶基因位于线性染色体上。在某些实施方案中,靶基因位于ecdna上。在一些实施方案中,靶基因位于第二ecdna上。在一些实施方案中,细胞是癌细胞。在一些实施方案中,细胞是癌前细胞。在某些实施方案中,细胞从包含癌细胞的多个细胞获得。在一些实施方案中,细胞从对象获得。在一些实施方案中,对象是以下中的至少一种:被诊断患有癌症、被怀疑患有癌症和处于患癌症的风险中。在一些实施方案中,细胞从细胞培养物获得。在一些实施方案中,细胞是脊椎动物细胞。在某些实施方案中,细胞是哺乳动物细胞,并且任选地是人细胞。在一些实施方案中,用于检测癌基因的水平和ecdna调节作用中的一种或更多种的手段包括直接测量调节ecdna和受调节癌基因之间的染色体间染色质接触频率。在一些实施方案中,用于检测癌基因的水平和ecdna调节作用中的一种或更多种的手段包括确定一种或更多种受ecdna调节的癌基因的转录水平,其中所述转录水平决定癌症的癌基因状态。在某些实施方案中,所述方法还包括在从包含所述癌症的第二多个细胞获得的癌细胞中重复步骤(a)和(b),并将在从第一多个细胞获得的癌细胞中检出的一种或更多种水平或作用分别与在从第二多个细胞获得的癌细胞中检出的水平或作用进行比较,其中所述水平和作用之一或二者的差异指示癌症的癌基因状态的变化。在一些实施方案中,所述方法还包括在确定第一多个癌细胞的癌症的癌基因状态之后并且在确定第二多个癌细胞的癌基因状态之前,将候选治疗剂与第二多个癌细胞接触,以及确定与候选治疗剂的接触对第二多个癌细胞的癌基因状态的影响。在一些实施方案中,第一和第二多个细胞从对象获得。在某些实施方案中,对象是以下中的一种或更多种:被诊断患有癌症、被怀疑患有癌症和处于患癌症的风险中。在一些实施方案中,第一和第二多个细胞从细胞培养物获得。在一些实施方案中,癌细胞是哺乳动物细胞,任选地是人细胞。在某些实施方案中,第一和第二多个细胞包含癌细胞。在一些实施方案中,所述方法还包括至少部分地基于所确定的癌症的癌基因状态来协助选择针对癌症的治疗。在某些实施方案中,所述方法还包括在癌细胞中鉴定受一种或更多种另外的ecdna调节的一种或更多种另外的癌基因,并确定所鉴定的一种或更多种另外的癌基因的水平和ecdna调节作用中的一种或更多种,以作为对癌症的癌基因状态的确定。
[0010]
根据本发明的另一方面,提供了鉴定细胞中染色体外dna(ecdna)的方法,所述方法包括使用染色质相互作用分析方法直接检测非线性dna分子与至少一个线性染色体之间的物理相互作用,其包括:(a)将细胞与固定剂接触,并对从细胞中分离的dna进行染色质邻近连接(chromatin proximity ligation);(b)进行染色质免疫沉淀;(c)从步骤(b)中免疫沉淀的dna生成文库;(d)对步骤(c)中生成的文库进行测序以生成测序数据;以及(e)分析步骤(d)中生成的测序数据以检测ecdna。在某些实施方案中,步骤(a)至(c)使用chia-pet或hi-c方法来进行。在一些实施方案中,步骤(b)包括在染色质免疫沉淀中使用抗rnapii抗
体。在一些实施方案中,步骤(c)和(d)包括分别使用配对末端标签和高通量测序。在某些实施方案中,所述方法还包括确定所检出的物理相互作用的频率。在一些实施方案中,所述方法还包括确定非线性dna分子的大小。在一些实施方案中,所述方法还包括确定细胞中非线性dna分子的拷贝数。在一些实施方案中,所述方法还包括在第一时间确定多个细胞中非线性dna分子的平均每细胞拷贝数,并将所确定的平均值与非线性dna分子的对照平均每细胞拷贝数进行比较。在某些实施方案中,非线性dna分子的对照平均每细胞拷贝数是在不同时间点在所述多个细胞中确定的非线性dna分子的平均每细胞拷贝数。在一些实施方案中,所述方法还包括确定非线性dna分子的至少一部分的序列。在一些实施方案中,所述方法还包括在所确定的序列中鉴定癌基因序列的存在。在某些实施方案中,细胞是癌细胞。在一些实施方案中,细胞是癌前细胞。在一些实施方案中,细胞从包含癌细胞的多个细胞获得。在某些实施方案中,细胞从对象获得。在一些实施方案中,对象是以下中的至少一种:被诊断患有癌症、被怀疑患有癌症和处于患癌症的风险中。在某些实施方案中,细胞从细胞培养物获得。在一些实施方案中,细胞是脊椎动物细胞。在一些实施方案中,细胞是哺乳动物细胞,并且任选地是人细胞。在某些实施方案中,所述方法还包括对多个细胞进行步骤(a)至(e)。
[0011]
根据本发明的又一方面,提供了选择治疗以在对象中降低癌症的方法,所述方法包括:在从对象获得的癌细胞中鉴定一种或更多种特定的受ecdna调节的癌基因的存在,并基于所鉴定的受ecdna调节的癌基因选择一种或更多种治疗。在一些实施方案中,鉴定包括上述鉴定细胞中染色体外dna(ecdna)的方法的任一个实施方案,所述方法包括:(a)检测非线性dna分子与染色体对中至少一个线性染色体之间的染色质相互作用,其中相互作用包括非线性dna分子与染色体对中至少一个线性染色体之间的接触,并且其中存在以下项则将非线性dna分子鉴定为ecdna:(i)在细胞中所检出的染色质相互作用的显著高频率;(ii)非线性dna分子与细胞中每个染色体对中至少一个线性染色体之间的接触;以及(iii)在多个细胞中非线性dna分子的平均每细胞拷贝数随时间的增加。在一些实施方案中,鉴定包括上述鉴定细胞中染色体外dna(ecdna)的方法的任一个实施方案,所述方法包括:使用染色质相互作用分析方法直接检测非线性dna分子与至少一个线性染色体之间的物理相互作用,其包括:(a)将细胞与固定剂接触,并对从细胞中分离的dna进行染色质邻近连接;(b)进行染色质免疫沉淀;(c)从步骤(b)中免疫沉淀的dna生成文库;(d)对步骤(c)中生成的文库进行测序以生成测序dna;以及(e)分析步骤(d)中生成的测序数据以检测ecdna。
附图说明
[0012]
图1a至b提供了图和描记(trace),其示出了ecdna特征可通过23对染色体上反式染色体相互作用频率(nstif)的分布来区分。图1a提供了在hf-3016和hf-3177ecdna(+)细胞系中所有23对染色体上由ecdna区域介导的反式相互作用的circos图。示出了ecmyc、ecegfr和eccdk4区域之间的紧密联系。图1b示出了ecdna(+)hf-3016和hf-3177细胞系以及ecdna(-)hf-3035系中全基因组归一化总和tif(nstif)以50-kb的区块大小(bin size)的分布。在7、8和12号染色体上观察到nstif升高。nstif沿全长7、8和12号染色体的分布如下所示,并且具有升高的nstif值的区域与已知的ecegfr、ecmyc和eccdk4区域匹配良好。
[0013]
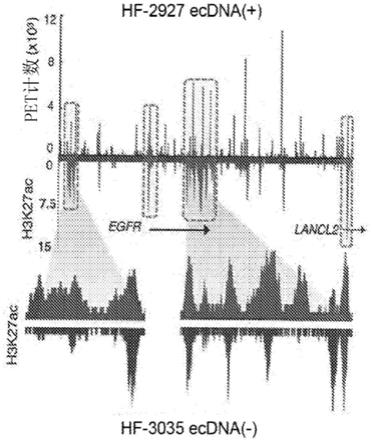
图2a至d示出了谱、图和箱形图(box plot),其示出了ecdna表现出对宽跨度的h3k27ac修饰的强烈增强的证据。图2a提供了与在hf-2354、hf-2927、hf-3016和hf-3177中
的各ecdna(+)细胞系中发现的ec-癌基因启动子相互作用的染色体非编码锚的
±
3kb内h3k27ac修饰富集谱。标记了在每个系中发现的锚的数目,并且每个区域显示为按在其相应细胞系中检出的信号强度排序的行(从顶部最高强度到最低强度)。图2b示出了在hf-2927中的ecegfr区域(chr7:54,929,292-55,441,765)的染色质相互作用频率和h3k27ac信号密度分布之间的一致性。下方的组:ecdna(+)hf-2927中ecegfr区域(上方)的h3k27ac信号密度谱。为了进行比较,示出了来自ecdna(-)hf-3035系中同一区域的h3k27ac信号密度谱(下图)。突出显示了示出信号强度和跨度的差异的区域。图2c至d示出了箱形图,其示出了h3k27ac峰关于来自四个ecdna(+)系中每一个的ecdna(a组,n=17、16、96和70)、其相应的反式相互作用染色体锚(b组,n=166、634、913和745)和其余的全基因组峰(c组,n=38,259、40,413、48,751和56,041)的倍数富集(图2c)和跨度大小分布(图2d)。在ecdna(-)hf-3035系中,a组(n=182)是指在全体ecdna等效区域中发现的h3k27ac峰,而c组(n=53,529)表示检出的其余全基因组峰。y轴在图2c和图2d中分别是log2和log
10
标度。中心线,中位数;箱体,第一和第三四分位数;影线(whisker),四分位距(interquartile range,iqr)的1.5倍;点,异常值。*:p值《0.005(单侧wilcoxon秩和检验)。对于每个成对比较,样品大小和精确p值。在图2c和图2d中的每一个中,x轴上从左到右的第一个a、b和c表示来自hf-2354的数据,第二个a、b和c表示来自hf-2927的数据,第三个a、b和c表示来自hf-3016的结果,第四个a、b和c示出了来自hf-3177的数据,而最后一个a和c示出了来自hf-3035的数据。
[0014]
图3a至c提供了维恩图、示意图和表格,其示出了ecdna介导的反式相互作用基因及其相关的相互作用网络。图3a至b示出了维恩图,其示出了四个ecdna(+)细胞系中每一个中的ecdna连接基因(图3a)和癌基因(图3b)的数目及其重叠。图3c示出了定义相互作用锚、节点、枢纽(hub)和群落(community)的过程流程(参见实施例部分中的方法)。将由染色质环连接的基因组区域定义为锚。将非重叠的锚合并为具有连接评分(连接节点数)的节点,其中将高连接度节点(≥平均连接评分+3s.d.)分类为枢纽。将具有广泛连接度的枢纽和节点共同定义为群落。总结了每个ecdna(+)系的与ecdna相关的群落、枢纽和癌基因的数目。
[0015]
图4a至b提供了对五种gbm来源神经球细胞系的chia-pet分析的总结。图4a示出了在来自ecdna(+)gbm来源细胞系的ecdna内扩增的基因的稳态表达水平。图4b示出了来自chia-pet数据的在五个细胞系中的每一个中检出的rnapii结合位点和长程(long-range)染色质相互作用的数目。将相互作用报告为三类,全基因组显著的顺式相互作用(pet计数≥3,p值《0.05且fdr《0.05,参见实施例部分,方法)、ecdna区域之间的相互作用(ecdna内相互作用)以及ecdna区域与23对线性染色体之间的相互作用(ecdna反式染色体)。仅报告了在两个锚处均检出的与rnapii结合位点的相互作用。
[0016]
图5a至c示出了示意图、分布和箱形显示结果,其表明通过chia-pet分析发现了ecdna特征。图5a为示意图,其示出了用于检测在ecdna中扩增的基因组区域及其相关染色质接触的染色质相互作用分析。进行rnapii chia-pet测定以捕获所有rnapii缔合染色质。ecdna携带活跃表达的癌基因,不受染色质区域的限制,并与其他染色体区域进行广泛接触,这可用于发现ecdna特定特征并表征其在活跃转录枢纽内的共调节基因。图5b示出了hf-2927和hf-2354中归一化的反式相互作用频率总和(nstif)以50-kb的区块大小在23对染色体中的分布。在其相应的细胞系中,7号染色体和8号染色体的相应的归一化nstif分布(在放大的nstif图中示出)揭示了预期的ecdna的位置分别包含egfr和myc。图5b,左侧示出
了全基因组2d染色质接触热图,其示出了染色体7p11和8q24上的区域的显著线对,表明与整个基因组的密切接触。示出了图5b右侧,其示出了在所有23对染色体上由ecegfr和ecmyc区域介导的反式染色体接触频率的circos图。图5c提供了箱形图,其示出了在四个ecdna(+)细胞系中已知ecdna区域和具有拷贝数增加≥3的染色体dna区域之间归一化的nstif。从左到右,n=31、3,423、11、5、15、82、25、833。ecdna区域中的nstif在统计学上高于具有拷贝数增加的区域中的nstif。对于hf-2354、hf-2927、hf-3016和hf-3177,p值(单侧wilcoxon秩和检验)分别为4e-22、4.5e-4、4.4e-10和7.1e-18。中心线,中位数;箱体,第一和第三四分位数;影线,四分位距(iqr)的1.5倍;点,异常值。
[0017]
图6a至c提供了热图,其示出了由基因组结构变体反映的染色质拓扑变化的chia-pet测定检测。图6a示出了chia-pet数据中通过一般染色质接触测量的空间染色质拓扑可通过2d接触热图进行可视化。示出了来自所有五种gbm患者来源神经球细胞系的2号染色体热图。图6b提供了在pten(上)以及cdkn2a和cdkn2b(下)分别缺失的hf-2927和hf-3035的基因组区域的2d接触热图。tad区域由蓝色线标出界线。基因座的缺失表现为染色质接触的丢失。图6c示出了由2d热图可视化为异常接触模式的dmd基因缺失、chr.3中的复杂重排和双易位t(3;6)的另外的结构变体。
[0018]
图7a至c示出了热图、circos图和示意图,其示出了ecdna被rnapii结合并介导广泛的染色体外内(intra-extrachromosomal)和反式染色体相互作用。图7a提供了ecdna(+)hf-2927和hf-2354与ecdna(-)hf-3035细胞系之间的2d接触热图比较。示出了相对于hf-3035中非ecdna egfr基因编码区,hf-2927中ecegfr区(chr7:54,860,254-55,535,856)的顺式相互作用和rnapii结合强度的谱;以及相对于hf-3035中非ecdna myc编码基因,hf-2354中ecmyc区域的两个片段(chr8:128,032,011-128,806,493和chr8:129,573,241-130,968,628)的顺式相互作用和rnapii结合强度的谱。图7b提供了hf-2927(左)和hf-3177(右)ecdna(+)细胞系中限定的ecdna区域的circos图。从内圈到外圈:ecdna内不同区域之间的ecdna内相互作用环;蓝色:ecdna内相互作用频率的分布;绿色:ecdna-染色体反式相互作用频率的分布;橙色(从外开始第三个环):h3k27ac倍数富集强度;棕色(从外开始第二个环):rnapii结合富集强度。信号轨迹(track)的分辨率为1kb。h3k27ac信号与相互作用频率之间的高一致性以灰色突出显示。图7c是示出了与来自ecdna及其染色体靶标的反式相互作用锚相关的基因组特征(启动子、基因间和基因内区域)的图。
[0019]
图8a至d提供了circos图、示意图和显微照片,其示出了ecdna区域显示出具有强h3k27ac富集的强烈的顺式和反式相互作用。图8a示出了hf-2354(左)和hf-3016(右)ecdna(+)细胞系中限定的ecdna区域的circos图。从内圈到外圈:ecdna的不同区域之间的ecdna内相互作用环;蓝色:ec内相互作用频率的分布;绿色:ecdna-染色体反式相互作用频率的分布;橙色,从外开始第三个环:h3k27ac倍数富集强度;棕色,从外开始第二个环:rnapii结合富集强度。信号轨迹的分辨率为1kb。高h3k27ac富集与相互作用频率之间的高一致性以灰色突出显示。图8b示出了ecdna连接的染色体相互作用锚(橙色)以及未连接与h3k27ac峰重叠的ecdna的锚(蓝色)的百分比。图8b左侧示出了基因内区域(g)的染色体锚。图8b右侧示出了基因间区域(i)(基因编码区域之外)的染色体锚。图8c示出了细胞特异性染色体h3k27ac峰与ecmyc启动子相互作用。以10-kb窗口示出了这些宽峰所在的基因组位置,其中hf-3177中的起始位置为chr3:42,090,000;chr5:148,938,000、chr1:224,353,000、chr1:
33,906,500、chr19:18,408,000、chr1:207,060,000;hf-3016中的起始位置为chr3:195,902,000、chr10:10,0120,000;且hf-2354中的起始位置为chr7:63,920,000。图8d提供了示出了来自hf-2927细胞的细胞中期染色体上h3k27ac免疫染色的图像。显著的dapi阳性染色体外点(extrachromosomal spot)与h3k27ac染色点重叠。
[0020]
图9a至e提供了箱形图和示意图,其示出了ecdna介导的染色质相互作用靶向癌基因用于空间聚集的亚核网络中的活跃转录。图9a示出了在来自四个ecdna(+)细胞系中每一个的与ecdna反式相互作用的染色体基因(n=1,887、1,270、1,483和1,157)与无反式染色体相互作用的基因(n=483、194、653和597)之间的rna表达(fpkm)的分布。*表示p值《0.005(单侧wilcoxon秩和检验)。对于每个成对比较,样品大小和精确p值。图9a在x轴上从左到右,第一个+、-示出了hf-2354的结果,第二个+、-示出了hf-2927的结果;第三个+、-示出了hf-3016的结果;第四个+、-示出了hf-3177的结果。图9b示出了染色体基因随ecdna接触频率的提高程度(0至9)的基因表达(fpkm)分布。对于每个ecdna (+)系,拟合值的95%置信区间以阴影显示。平滑的fpkm以实心的拟合线表示。图9b,每个反式相互作用频率(1至10)的每组四个箱体从左到右示出了hf-2354、hf-2927、hf-3016和hf-3177的结果。图9c是示出了ecdna连接的癌基因(n=87、56、78和54)与全转录组(n=21,186、18,988、19,206和19,180)的表达水平的箱形图。通过单侧wilcoxon秩和检验的成对比较的p值为1.2e-7、1.4e-5、9.0e-8和1.1e-5。图9a至c,中心线,中位数;箱体,第一和第三四分位数;影线,四分位距(iqr)的1.5倍;点,异常值。图9c在x轴上从左到右,onco、野生型(wt)示出了hf-2354的结果,第二个onco、wt示出了hf-2927的结果;第三个onco、wt示出了hf-3016的结果;第四个onco,wt示出了hf-3177的结果。图9d是示出了癌基因通过ecdna介导的染色质相互作用聚集在空间近端(proximity)内的图。示出了在hf-3016和hf-3177中由ecdna连接枢纽介导的两个群落的实例。节点之间的反式相互作用以棕褐色线表示,粗细用log
10
(ipet计数)表示。蓝色圆圈是基因(启动子)节点,紫色圆圈是注释为癌基因的基因(启动子)节点,灰色圆圈是基因间节点。除了手动调整为较小尺寸的ecdna圆圈之外,圆圈的尺寸与连接评分(整个全基因组网络中的边数)成比例。标记选定的基因以示出其在群落之间的变异性。图9e是模型的示意图,该模型示出了ecdna作为移动增强子发挥作用来进行广泛的顺式和反式染色体相互作用以将癌基因募集到活跃转录枢纽中并促进癌细胞中全局转录扩增。
[0021]
图10a至c提供了图、示意图和箱形图,其示出了ecdna反式连接基因被活跃地转录。图10a示出了在从五种gbm来源神经球细胞系测量的所有表达的rna之间的高相关性,代表了rna-seq分析的一致性。图10b示出了基于其ecdna连接状态,将基因分为三个不同的类别;组i:具有与ecdna连接的启动子的基因,组ii:具有与其他启动子反式连接但没有ecdna连接的启动子的基因,和组iii:不具有反式相互作用的基因。图10c提供了在hf-2354、hf-2927、hf-3017和hf-3177ecdna(+)细胞系中来自组i、ii和iii的基因的稳态rna表达(fpkm)的箱形图。*表示基于单侧wilcoxon秩和检验的显著p值。确定了成对比较的精确p值,并将基因分类在组i、ii和iii中的每一个中。
具体实施方式
[0022]
本发明部分地涉及鉴定染色体外环状dna(ecdna)以及ecdna在疾病例如癌症中的作用的方法。本发明的某些方法包括表征ecdna及其在癌症基因组中的致癌性改变。现已确
定了染色质相互作用测定(例如但不限于chia-pet染色质相互作用测定)可用于推进ecdna的鉴定,并表征在功能上影响疾病(例如但不限于癌症)中转录程序的全基因组ecdna介导的染色质接触。使用胶质母细胞瘤患者来源神经球培养物中的ecdna进行了研究,其中一些在本文中进行了描述。在这些研究中,通过存在ecdna的广泛的染色体间相互作用来鉴定ecdna。ecdna-染色质接触焦点的特点是主要集中在染色体启动子上的广泛且高水平的信号,表明了在染色体基因转录的全基因组激活中的主要调节作用。在本发明的一些实施方案中,信号包括h3k27ac信号。对ecdna的染色体靶标的破译揭示了与在空间上聚集在ecdna-染色质连接网络中的活跃表达的癌基因的相关性。所进行的研究结果表明,ecdna除了癌基因扩增的表现之外还可作为移动转录扩增元件发挥作用以激活癌症中的癌基因表达。
[0023]
鉴定ecdna
[0024]
为了鉴定ecdna的染色质组织以及该组织如何促进基因转录调节,采用染色质相互作用测定来检查和询问相同细胞系上的一般空间染色质组织以及蛋白质因子介导的长程染色质相互作用二者。可用于本发明的一些实施方案中的染色质相互作用测定的一些非限制性实例是:chia-pet方法、chip方法和hi-c方法。现已表明,已知的ecdna可通过其强烈和异常的分子内和分子间全基因组染色质接触来鉴定。此外,为破译rna聚合酶ii(rnapii)介导的ecdna连接组及其染色体配偶体(chromosomal partner)而进行的研究导致鉴定了ecdna与活跃表达的常染色体癌基因之间的相关性。该发现表明,ecdna的机制作为促进肿瘤进展的移动转录增强子来发挥作用。
[0025]
除了提供癌症基因组中ecdna靶向染色质相互作用组的详细表征之外,本文中公开的染色质相互作用测定的使用提供了有效的手段来精确定位ecdna内扩增的基因组结构域,这是基于其在ecdna内和ecdna之间以及在ecdna和线性dna之间的密切染色质接触进行的。试图表征ecdna所使用的现有方法利用了基于成像的分析或全基因组测序和dna拷贝数数据的计算分析。本文中公开的本发明的一些实施方案与依赖于对具有拷贝数增加的区域进行的结构分析或显微成像方法的现有方法的不同之处至少在于本文中提供的方法可用于通过染色质相互作用测定(例如如但不限于:chia-pet和hi-c)直接测量染色体间染色质接触频率。本发明方法的一些实施方案提供了可用于鉴定一个或更多个ecdna特征的无偏方法,所述ecdna特征例如但不限于:ecdna大小、不同ecdna之间的大小比较、ecdna拷贝数;ecdna序列信息和ecdna序列背景。另外,本发明方法的一些实施方案可用于确定和/或评估一个或更多个特征,例如但不限于:ecdna分子和染色体dna分子的不同区域之间的接触频率和模式。在本发明的方法中染色质相互作用分析的使用提供了对ecdna分子物理结构和连续性的了解。
[0026]
本发明的某些方面包括在一个细胞或多个细胞中鉴定一种或更多种ecdna的方法。所述方法可包括检测非线性dna分子与至少一个线性染色体之间染色质相互作用的手段。在本发明的一些实施方案中,所述方法包括检测非线性dna分子与染色体对中至少一个线性染色体之间的染色质相互作用。本文中使用的术语“检测染色质相互作用”意指检测染色质相互作用的频率、相互作用中的ecdna、相互作用中的靶基因和染色质相互作用的其他特征中的一个或更多个。染色质相互作用的特征可包括但不限于以下一个或更多个:非线性dna的大小和细胞中非线性dna分子的拷贝数等。在一些实施方案中,本发明的方法包括
将染色质相互作用的一个或更多个特征进行比较,例如,在第一确定时间确定多个细胞中非线性dna分子的平均每细胞拷贝数,并将所确定的平均值与非线性dna的对照平均每细胞拷贝数进行比较。在本发明的一些实施方案中,将染色质相互作用的一个或更多个特征进行比较可包括在第一确定时间确定多个细胞中非线性dna分子的平均每细胞拷贝数,并将所确定的平均值与非线性dna的对照平均每细胞拷贝数进行比较。应理解,在本发明的一些实施方案中,非线性dna分子的对照平均每细胞拷贝数是在与另一确定时间点不同的时间点在所述多个细胞中确定的非线性dna分子的平均每细胞拷贝数。检测染色质相互作用的特征的另一些非限制性实例包括:确定非线性dna的至少一部分的序列,并在所确定的序列中鉴定癌基因序列的存在。
[0027]
现已鉴定了可用于鉴定细胞中ecdna的特定特征。特征通过细胞中染色质相互作用的一个或更多个所确定的特征来鉴定。例如,将染色质相互作用鉴定为包含ecdna与至少一个线性染色体之间的接触包括:鉴定(i)细胞中所检出的染色质相互作用的显著高频率;(ii)非线性dna分子与至少一个线性染色体(例如但不限于细胞中每个染色体对中至少一个线性染色体)之间的接触;以及(iii)多个细胞中非线性dna分子的平均每细胞拷贝数随时间的增加。在本发明的一些实施方案中,这三个特征(特征i至iii)的存在将非线性dna分子鉴定为ecdna。
[0028]
现已鉴定了ecdna的数个特征,并且在本发明方法的一些实施方案中,细胞中非线性dna分子中特征的存在证实了非线性dna分子被鉴定为ecdna。本发明方法的某些实施方案包括确定以下特征中的1、2或3个,作为确认所检出的非线性dna被鉴定为ecdna的手段。现已鉴定的ecdna的一个这样的特征是,包括ecdna及其靶基因的染色质相互作用以比细胞中其他类型的染色质相互作用显著更高的水平和频率发生。本文中使用的术语所检出的染色质相互作用的“显著高频率”意指所检出的这样的染色质相互作用的数目在统计学上显著高于如果染色质相互作用不包括ecdna时所检出的染色质相互作用的数目。
[0029]
现已鉴定的ecdna的另一个特征是存在ecdna与细胞中至少一个线性染色体之间的接触。在本发明的一些实施方案中,ecdna特征包括存在ecdna与细胞中每个染色体对中至少一个线性染色体之间的接触。例如,尽管不旨在进行限制,但将被鉴定为与细胞中至少一个染色体接触的非线性dna分子鉴定为ecdna。在另一个非限制性实例中,将被鉴定为与细胞中每个染色体中(例如,在人二倍体细胞中的23对染色体的每一对中)的至少一个染色体接触的非线性dna分子鉴定为ecdna。在后一个实例中,将存在ecdna与位于人细胞中23对染色体中每一对中的至少一个线性染色体上的基因靶标的相互作用。
[0030]
现已鉴定的ecdna的第三个特征是ecdna的平均每细胞拷贝数随时间增加。因此,作为一个非限制性实例,在从细胞群获得的细胞样品中,确定了非线性dna拷贝数的平均值。稍后,从细胞群获得第二细胞样品,并确定非线性dna拷贝数的平均值,并将其与第一样品中确定的平均数进行比较。后一样品中非线性dna的平均数目的增加支持了非线性dna是ecdna的结论。在本发明的一些实施方案中,平均数目的增加可以是增加至少1%、2%、3%、4%、5%、6%、7%、8%、9%、10%、15%、20%、25%、30%、35%、40%、45%、50%、55%、60%、65%、70%、75%、80%、85%、90%、95%、100%、125%、150%、200%、250%或500%,包括所述范围内的所有百分比。在本发明的一些实施方案中,平均数目的增加可以是增加至少500%、1000%、1500%、2000%或5000%。
[0031]
在本发明的一些实施方案中,细胞或多个细胞是在两个或更多个不同时间点从样品、培养物或对象获得的。获得两个细胞样品之间的时间长度可基于包括但不限于以下因素独立选择:对象的便利性、健康护理提供者的便利性、癌症的状态或阶段、癌症的发展速度、肿瘤生长速度等。在本发明的一些实施方案中,获得两个细胞样品之间的时间间隔为至少1、2、3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、21、22、23、24、25、26、27、28、29和30天。在本发明的一些实施方案中,获得两个细胞样品之间的时间间隔为至少1、2、3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、21、22、23、24、25、26、27、28、29、30、31、32、33、34、35、36、37、38、39、40、41、42、43、44、45、46、47、48、49、50、51和52周。在本发明的一些实施方案中,获得两个细胞样品之间的时间间隔为至少1、2、3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、21、22、23、24、25、26、27、28、29、30、31、32、33、34、35、36、37、38、39、40、41、42、43、44、45、46、47、48或更多个月。在本发明的一些实施方案中,获得两个细胞样品之间的时间间隔为至少1、2、3、4、5、6、7、8、9、10或更多年。应理解,可收集多于两个细胞样品用于本发明方法的某些实施方案,并且获得两个样品之间的时间间隔可独立地选择,并且可以但不必与获得细胞样品的其他时间间隔相同。
[0032]
ecdna和靶癌基因
[0033]
本文中所述的工作确认了ecdna与靶基因(其的一个非限制性实例是癌基因)之间的调节关系。本文中使用的术语“靶癌基因”意指其活性受ecdna调节的癌基因。调节靶癌基因转录的ecdna可通过基因调节系统(gene regulatory system,grs)进行调节。grs包括ecdna中的基因调节子。ecdna基因调节子可以是基因激活子,或在一些情况下可以是基因沉默子。应理解,ecdna与其靶基因之间的相互作用可包括ecdna基因调节子序列与转录因子的结合,所述转录因子还结合靶癌基因的基因调节元件。转录因子与基因调节元件(其包含dna的特定短区域)的结合刺激靶癌基因的转录。
[0034]
结合ecdna基因调节子和靶癌基因的转录因子可包含多肽复合物并充当ecdna基因调节子和靶癌基因的基因调节元件之间的“连接子(connector)”。术语“基因调节元件”意指负责和/或参与靶癌基因转录的dna序列,例如但不限于启动子或增强子序列。本文中使用的ecdna基因调节子与其靶癌基因之间的相互作用包括ecdna基因调节子与转录因子的接触,所述转录因子还接触靶癌基因的基因调节元件,例如靶癌基因的启动子序列。ecdna/癌基因相互作用增强靶癌基因的转录,这可导致或促进癌症的发展。在一些实施方案中,癌症可存在于包含ecdna及其靶癌基因的细胞中。由ecdna/癌基因相互作用产生的癌细胞可存在于对象中。不同的靶癌基因可由于ecdna/癌基因相互作用而具有增强的转录,并且细胞可包含一个、两个或更多个不同的癌基因,其转录因ecdna/癌基因相互作用而提高。对象中的两种或更多种细胞可包含相同的靶癌基因或可包含不同的靶癌基因,其转录受一种或更多种ecdna调节。对象的癌症可通过一个、两个或更多个不同癌基因的活性而产生和/或维持,其中每个癌基因的转录受一种或更多种ecdna调节。
[0035]
在对象的癌症中两个或更多个不同的癌基因被激活的情况下,针对不同癌基因和/或不同的ecdna/癌基因相互作用的两种或更多种不同的癌症治疗可用于有效治疗对象的癌症。在本发明的一些实施方案中,可至少部分地基于ecdna与特定癌基因之间的相互作用的存在或不存在以及通过相互作用对该癌基因的调节来选择和/或向对象施用癌症治疗。
[0036]
除了可用于鉴定ecdna的本发明方法之外,本发明的某些方法可用于通过评估ecdna与其靶癌基因之间相互作用的存在或不存在来评估细胞中癌症的状态。这些方法部分地基于对三维基因组组织及其在基因调节中的作用的改进理解。本发明的某些实施方案包括鉴定ecdna与细胞中至少一个靶癌基因之间的相互作用的方法。另外,本发明方法的某些实施方案可用于评估ecdna/癌基因相互作用的频率和效果。在本发明的一些实施方案中,ecdna与其靶癌基因的相互作用增强(在本文中也被称为“增加”)靶癌基因的转录。
[0037]
染色质相互作用测定
[0038]
本发明的某些方面包括使用染色质相互作用测定。在本发明方法的某些实施方案中,使用染色质相互作用测定来确定染色体的结构特征、鉴定细胞中的ecdna以及确定ecdna与靶基因(例如但不限于癌基因)之间的相互作用。本文中公开的染色质相互作用测定和分析还允许确定ecdna对靶癌基因的转录调节。
[0039]
测定细胞中染色质相互作用的手段的一个非限制性实例是通过配对末端标签测序进行的染色质相互作用分析(chromatin interaction analysis by paired-end tag sequencing,chia-pet),它将chip与染色质构象捕获(chromatin conformation capture)(3c)技术相结合(参见fullwood,et al.2009,nature,vol.426(7269):58-64;其内容通过引用整体并入本文)。chia-pet方法允许检测远距离dna区域之间的相互作用,这些远距离dna区域通过目的蛋白质或蛋白质复合物与彼此进行相互作用。在chia-pet方法的一个非限制性实例中,使用针对目的蛋白质的抗体将来自细胞的染色质交联、消化并牵出(pull down)。将接头序列与dna的末端连接,并且接头序列的存在促进了其与彼此的连接(zhang et al.,2012,methods vol.58,no.3:289-299;其内容通过引用整体并入本文)。这导致来自基因组的两个不同区域的杂交dna片段。对生成的文库进行测序,并且结果鉴定了与彼此以及目的蛋白质相互作用的dna区域。chia-pet先前已用于定位转录因子的相互作用,并且其在本发明方法的一些实施方案中的使用允许鉴定ecdna、ecdna的靶基因,以及通过ecdna/靶基因相互作用对靶基因转录的调节。使用chia-pet的染色质相互作用分析现已用于染色质相互作用的全基因组发现。现有的可视化方法和计算方法通常不适合检测弱相互作用或动态相互作用,而这一缺点可通过使用chia-pet方法来补救。
[0040]
用于本发明的某些实施方案中的鉴定和评估染色质相互作用的手段的另一个非限制性实例是hi-c评估方法。基于hi-c的方法可用于本发明的一些实施方案,部分原因在于其提供能够测量任何两个给定基因组基因座之间的染色质相互作用强度的无偏全基因组覆盖的能力。在本发明的某些实施方案中,hi-c数据可用于评估全基因组染色质组织,例如拓扑相关结构域(topologically associating domain,tad)、在3-d空间中相关联的基因组的线性连续区域。多种本领域已知的算法常规地用于从hi-c数据中鉴定tad(参见例如dixon et al,2012nature 485(7398):376-80;其内容通过引用整体并入本文)。
[0041]
以下是hi-c分析中要素的一般概述。细胞的基因组是交联的,这保留了基因组基因座之间的相互作用。有许多本领域已知的固定方法适用于在hi-c方法中交联细胞基因组。交联之后,使用限制酶切割交联的基因组,所得片段的大小决定了通过hi-c方法进行的相互作用定位的分辨率。可使用的限制酶的一些非限制性实例是每4000bp进行切割、在人基因组中产生约100万个片段的那些(例如ecor1或hindiii)。对于更高分辨率的相互作用定位,也可使用更频繁地进行切割的限制酶。在消化步骤之后,这些片段在有利于交联的相
互作用片段之间的连接而不是非交联片段之间的连接的条件下随机连接。然后可通过使用例如但不限于聚合酶链反应(polymerase chain reaction,pcr)的方法扩增连接的连接点来量化相互作用的基因座,参见例如,naumova,et al.,2012methods.58(3):192-203和gavrilov,et al.,2013plos one.8(3):e60403,其各自的内容通过引用整体并入本文)。某些hi-c方法可包括高通量测序以发现片段的核苷酸序列,例如ecdna的序列。
[0042]
本文中描述了可包括在用于本发明的实施方案中的chia-pet和/或hi-c方法中的另外的步骤和程序。例如,计算方法、分析方法、数据评估方法等。除了染色质相互作用分析的chia-pet和hi-c相关方法之外,其他染色质相互作用测定和分析方法是本领域已知和使用的并且适合用于本发明方法的一些实施方案中。另外的染色质相互作用测定/分析方法的一些非限制性实例包括:4c(nat genet.2006nov;38(11):1348-54)、hi-c(lieberman-aiden,e.et al.326,289-293(2009))、捕获hi-c(nat genet.2015jun;47(6):598-606)、plac-seq(cell research.2016;26(12):1345-8)和hichip(nat methods.2016nov;13(11):919-922);其各自的内容通过引用整体并入本文。
[0043]
相互作用分析——一般信息和非限制性实施方案
[0044]
应理解,相互作用分析的以下描述并非旨在进行限制,而是旨在举例说明使用本发明方法的实施方案获得的结果和信息。其他特定细胞类型、区域、癌基因等可用于本发明的一些实施方案中。在本文中的实施例部分中示出了关于ecdna与其靶癌基因之间相互作用的分析的另外的信息。这样的信息包括但不限于:本发明方法的一些实施方案用于使用chia-pet数据、chip-seq文库构建和数据分析等发现已知和未知ecdna区域的示范用途。
[0045]
本发明方法的某些实施方案包括染色质接触程度的量化。在一个非限制性实例中,使用描述了在所有23对染色体中归一化的全基因组反式相互作用频率(nstif)的度量进行量化。将ecdna区域鉴定为在具有ecdna的区域中具有高度升高的nstif水平。另外,与ecdna片段连接的高nstif区域显示出在整个基因组中的反式接触,表明来自染色体外遗传元件的动态ecdna连接度。在该实例中观察到的全基因组接触模式是由ecdna的移动性质造成的。在该非限制性实例中,进行了验证步骤以验证所鉴定的高nstif对ecdna的染色体外性质具有特异性。在该实例中,结果证实了ecdna在整个基因组中接触频率的升高不仅仅是dna剂量作用的结果,而是由其自主能力所决定。
[0046]
在另一个非限制性实例中,在ecdna的基因组区域内检测到高频率的顺式相互作用。在hf-2927ecdna(+)细胞中,在约530kb ecdna区域内观察到的顺式相互作用强度为2,879,与在ecdna(-)hf-3035中的相同区域中的仅12相比,为其240倍高。高接触提高直接反映了该ecegfr的大小和基因组结构二者。类似地,结果表明hf-2354中限定的ecmyc区域的两个片段内和两个片段之间的强烈的顺式相互作用。广泛的rnapii拴系(tethered)染色质接触(定义为在通过相互作用连接的dna区域检出的rnapii结合,被称为锚)以在ecdna内不同区域之间(被称为ecdna内)的顺式作用以及与线性染色体上的其他基因或调节元件的反式作用(被称为反式相互作用)被检测到。已鉴定,染色体外内连接模式显示出具有高频率相互作用和强烈接触的焦点的不同环对,预计其共同来源于不同ecdna分子之间的接触和单个ecdna内的折叠。在ecdna与其染色体配偶体之间的反式相互作用中,将ecdna上的锚鉴定为主要在基因内或基因间非编码区中,而其反式相互作用染色体锚主要位于启动子处。这种相互作用的并置支持了这些接触的转录功能。
[0047]
在评估ecdna与转录调节区域相互作用的一个非限制性实例中,进行h3k27ac谱分析以标记活性增强子和启动子。在ecdna上扩增的癌基因的调节通过评价其反式染色体相互作用区域来检查。这些ecdna连接非编码染色体锚表现出与h3k27ac峰高度重叠;其显著高于来自无ecdna接触的反式相互作用非编码染色体锚的那些,这支持了以下结论:通过染色质接触在线性染色体上接合增强子进一步增强了ecdna上癌基因的转录。
[0048]
在本发明的某些实施方案中,ecdna相互作用通过观察ecdna内高频率接触焦点与h3k27ac峰之间的共同出现来评估,结果支持了这些相互作用锚的表现类似于活性增强子的结论。在相互作用评估方法的该非限制性实例中,530kb ecegfr区域内的h3k27ac峰与hf-2927中的高相互作用频率区域共对齐(co-align),并且当与ecdna (-)细胞(hf-3035)中染色体egfr区域中的h3k27ac峰相比,表现出作为在更广泛的基因组跨度下密切接近的簇的模式,支持了增强子信号在ecdna的染色质接触位点上积累的结论。在这个实例中,使用靶向h3k27ac的抗体针对中期hf2927细胞进行的免疫染色表现出h3k27ac与标记ecdna的dapi信号之间的重叠信号,这确定了增强子功能与ecdna之间的相关性。
[0049]
在本发明的某些实施方案中,方法包括定量评估与ecdna介导的反式染色质相互作用相关的h3k27ac信号的提高,并且发现与ecdna染色质相互作用锚相关的h3k27ac峰与无ecdna接触的全基因组h3k27ac峰的那些相比具有显著更高的富集。在评估的该非限制性实例中,确定了h3k27ac信号的强烈增强是ecdna所特有的。
[0050]
在使用本发明方法的另一个非限制性实例中,使用所述方法评价了在让人联想到“超级增强子(super enhancer)”的ecdna中观察到的增强子特征。在该非限制性实例中,对在ecdna及其反式相互作用染色体锚中检出的h3k27ac峰的跨度大小进行了检查。发现ecdna上的h3k27ac峰与无ecdna接触的染色体h3k27峰相比具有显著更长的跨度。序列分析显示对调节rnapii一般转录和细胞增殖至关重要的转录因子(包括jun、fos和atf)结合基序的富集。总的来说,rnapii信号的顺式和反式会聚与h3k27ac信号的强烈增强支持了以下结论:ecdna分子能够广泛地连接整个基因组中的rna聚合酶机构,确定了作为全基因组转录放大器的功能。
[0051]
在一个非限制性实例中,本发明的方法用于确定与ecdna反式相互作用相关的增强子信号的提高是否导致活性转录。在该实例中,检查了rna表达,并且结果表明,与无其他反式染色体接触的基因或与ecdna无接触但与其他基因具有反式染色体相互作用的基因相比,ecdna相互作用基因具有显著更高的表达水平。此外,在该实例方法中,ecdna连接基因的表达水平被确定为与其ecdna接触的频率(通过独立的反式相互作用的数目来测量)呈正相关,这支持了以下发现:ecdna连接与转录活性和高度增强的h3k27ac特征高度相关,表明ecdna可充当全局转录扩增机构。
[0052]
除了募集单独癌基因之外,ecdna还被确定为焦点,在哪里许多癌基因通过其与ecdna的相互作用而聚集在一起呈空间接近。这些ecdna连接的癌基因中的许多驻留在每个染色质网络中,这支持了以下结论:癌基因的共聚集是ecdna采用的基于结构的机制,以实现协调的转录共激活来促进肿瘤发生。
[0053]
评估癌症状态
[0054]
现已表明,ecdna可通过染色质相互作用增强染色体外和染色体基因转录。本发明的一些实施方案包括在细胞中鉴定ecdna的方法。本发明的某些实施方案提供了确定ecdna
对靶基因(例如其转录受一种或更多种ecdna调节的癌基因)的作用的方法。已确定ecdna可通过染色质相互作用来增强染色体外和染色体基因转录的表达。该发现与ecdna的普遍性和多样性相组合,确定了ecdna、ecdna/靶癌基因相互作用和靶基因作为在疾病(例如癌症)中的治疗性干预的靶标。本发明的方法部分地基于肿瘤进化中遗传结构与表观遗传结果之间的相互作用的鉴定。
[0055]
本发明方法的一些实施方案提供了鉴定ecdna、鉴定ecdna与靶基因的相互作用以及ecdna与靶基因相互作用的影响的手段。ecdna活性在癌症中的作用以及这种染色体外结构的独特基因组动力学为靶向ecdna及其激活的染色体靶基因和激活的ecdna靶基因提供了新方法,例如用于治疗应用。
[0056]
建立并负责细胞状态(status)或状况(state)的基因表达程序包括但不限于结合ecdna基因调节子和靶癌基因的基因调节子元件的一种或更多种转录因子的活性。特定基因组元件的一些非限制性实例是增强子元件,其结合转录因子并可使长距离成环以接触和调节特定基因。尽管ecdna基因调节子序列与基因调节元件(例如但不限于靶癌基因的启动子)之间的相互作用,现已就ecdna与线性染色体上的基因调节元件之间的相互作用进行了研究。本发明的某些方面可用于获得染色体外dna(ecdna)的身份信息,所述染色体外dna(ecdna)通过其与线性基因组元件(例如癌基因启动子)的相互作用来参与细胞基因调节过程。另外,本发明的某些方法可用于评估参与细胞基因表达程序(在一些情况下包括异常基因表达程序,例如存在于癌细胞中的那些)的两种或更多种ecdna之间的调节相互作用。
[0057]
确定候选治疗剂并选择癌症的治疗
[0058]
在本发明的一些方面中,提供了在癌细胞中鉴定一种或更多种癌基因状态的方法。本发明的方法可用于确定一种或更多种靶癌基因的转录水平,其中一种或更多种靶癌基因的水平升高确定了癌症的可能性。在本发明的一些实施方案中,多种癌细胞可以是获得用于比较研究和测试候选治疗的细胞的来源。例如,尽管不旨在进行限制,但多种癌细胞可以是培养物中或对象中的癌细胞并且可以维持在相同环境中。在本发明的一些实施方案中,来自这样的培养物或对象的一种或更多种癌细胞包括在本发明的方法中以评估细胞对于ecdna/癌基因相互作用的状态。将不同的一种或更多种癌细胞与治疗剂或候选治疗剂接触并且将经接触的细胞包括在本发明的方法中以评估细胞对于ecdna/癌基因相互作用的状态。在未经接触和经接触癌细胞中确定的ecdna/癌基因相互作用可被确定并彼此比较或与适当的对照进行比较,提供关于治疗或候选治疗对ecdna/癌基因相互作用和癌症状态的作用的信息。
[0059]
当用于提及癌细胞时,本文中使用的术语“状态”意指一种或更多种特定ecdna/癌基因相互作用的存在或不存在。例如,尽管不旨在进行限制,但癌症的初始状态可包括ecdna与癌基因a和b之间的相互作用,并且随着癌症进展,其状态可被确定为包括ecdna与癌基因a、b和c之间的相互作用。
[0060]
在本发明的一些实施方案中,鉴定受ecdna调节的癌基因提供了可用于帮助选择对患有癌症的对象的治疗的信息。在一些实施方案中,对象可针对癌症的预倾向性(predisposition)或对对象中存在或怀疑存在的癌症进行分期来筛选。本发明方法的一些实施方案可用于筛选对象中的癌症或癌症状态,并且这样的方法可包括以下中一种或更多种:在细胞中鉴定ecdna/癌基因相互作用以及确定ecdna对靶癌基因的调节作用。这样的方
法可用于鉴定对象的癌症状态。另外,本发明的方法可用于评估候选药剂在癌症中对ecdna对其靶癌基因的调节作用的作用,并且评估结果可用于帮助选择针对癌症的治疗。
[0061]
本发明方法的一些实施方案可用于在细胞、组织、对象和多种细胞(或细胞群)中的一种或更多种中评估癌症状态。本文中使用的术语“癌症”用于提及恶性赘生物。一些示例性癌症包括但不限于听神经瘤;腺癌;肾上腺癌;肛门癌;血管肉瘤;阑尾癌;胆道癌(biliary cancer)(例如,胆管癌(cholangiocarcinoma));膀胱癌;乳腺癌(例如,乳腺腺癌、乳腺乳头状癌、乳腺癌、乳腺髓样癌);脑癌(例如,脑膜瘤、胶质母细胞瘤、胶质瘤(例如,星形细胞瘤、少突神经胶质瘤)、髓母细胞瘤);宫颈癌(例如,宫颈腺癌);结直肠癌(例如,结肠癌、直肠癌、结直肠腺癌);结缔组织癌;上皮癌;室管膜瘤;内皮肉瘤(例如,卡波西肉瘤(kaposi'ssarcoma)、多发性特发性出血性肉瘤);子宫内膜癌(例如,子宫癌、子宫肉瘤);食管癌(例如,食管腺癌、巴雷特腺癌(barrett’sadenocarcinoma));尤因肉瘤(ewing's sarcoma);眼癌(例如,眼内黑素瘤、视网膜母细胞瘤);家族性嗜酸性粒细胞增多症(familiar hypereosinophilia);胆囊癌;胃癌(例如,胃腺癌);胃肠癌;头颈癌(例如,头颈鳞状细胞癌、口腔癌)、喉癌;造血系统癌症(hematopoietic cancer)(例如,白血病,例如急性淋巴细胞白血病(acute lymphocytic leukemia,all);淋巴瘤,例如霍奇金淋巴瘤(hodgkin lymphoma,hl)和非霍奇金淋巴瘤(non-hodgkin lymphoma,nhl);多发性骨髓瘤(multiple myeloma,mm);血管母细胞瘤;肾癌(例如,肾母细胞瘤,亦称维尔姆斯瘤(wilms'tumor),肾细胞癌);肝癌(例如,肝细胞癌(hcc),恶性肝癌);肺癌(例如,支气管肺癌、小细胞肺癌(small cell lung cancer,sclc)、非小细胞肺癌(non-small cell lung cancer,nsclc)、肺腺癌);平滑肌肉瘤(leiomyosarcoma,lms);肥大细胞增多症(例如,系统性肥大细胞增多症);恶性间皮瘤;肌肉癌;骨髓增生性病症(myeloproliferative disorder,mpd);神经母细胞瘤;神经纤维瘤;神经内分泌癌;骨肉瘤;卵巢癌;乳头状腺癌;胰腺癌;阴茎癌;前列腺癌;直肠癌;横纹肌肉瘤;唾液腺癌;皮肤癌;黑素瘤;小肠癌;软组织肉瘤;皮脂腺癌;小肠癌;汗腺癌;滑膜瘤;睾丸癌;甲状腺癌;尿道癌;阴道癌;和外阴癌。
[0062]
癌症可以是原发性癌症或转移性癌症,并且可被认为是早期或晚期癌症,或者对象的癌症分期可用本领域已知和常规实践的一种或更多种癌症分期分类来表征。在本发明的一些方面中,癌症是对象中的第一种癌症,并且在本发明的某些方面中,癌症可以是先前癌症的复发或再现。在一些情况下,本发明方法的一个实施方案可用于评估未曾用癌症治疗进行治疗的对象的癌症状态。在某些实施方案中,本发明的方法用于评估已经用或目前正在用一种或更多种癌症治疗进行治疗的对象的癌症状态。癌症治疗的一些非限制性实例包括:手术、放射治疗、化学治疗、免疫治疗、饮食治疗或其他本领域已知的治疗方法。
[0063]
本发明的某些实施方案包括帮助确定和/或选择用于对象的一种或更多种治疗方案的方法。例如,尽管不旨在进行限制,但本发明的一些实施方案可用于至少部分地基于在从对象获得的癌细胞中鉴定的ecdna/癌基因相互作用的状态来帮助选择用于对象的癌症的治疗。使用本发明方法的一个实施方案确定对象的癌症状态基于所鉴定的ecdna/癌基因相互作用允许选择一种或更多种治疗。例如,尽管不旨在进行限制,但本发明的方法可用于通过在从对象获得的癌细胞中鉴定一种或更多种ecdna/靶癌基因相互作用来检测对象的癌症状态。本发明的方法还可用于在所检出的ecdna/相互作用中鉴定一种或更多种特定癌基因和其他组分,并且该信息可用于帮助选择用于对象的癌症的治疗。例如,如果在来自对
象的癌细胞中检出ecdna与癌基因a和癌基因b之间的相互作用,则该信息可帮助选择针对癌症的导致以下中一种或更多种的治疗:(i)降低ecdna与癌基因a的相互作用和(ii)降低ecdna与癌基因b的相互作用。基于使用本发明方法的一个实施方案确定的ecdna/癌基因相互作用信息,可通过特定癌基因/ecdna相互作用对对象的癌症进行分类并且可选择适当的治疗并将其施用于对象以降低特定的癌基因/ecdna相互作用。
[0064]
在提供的本发明方法的某些实施方案中,允许确定施用于癌细胞或者患有癌症、怀疑患有癌症或处于提高的患癌症的风险中的对象的癌症治疗的效力。在一个非限制性实例中,本发明方法的一个实施方案用于在从对象获得的癌细胞中确定癌症的初始状态。癌症状态被确定为包括一种或更多种ecdna与癌基因a、癌基因b和癌基因c之间的鉴定的相互作用。至少部分地基于ecdna与三种癌基因的所鉴定相互作用来选择用于对象的癌症治疗。在向对象施用选择的治疗之后,本发明的方法用于治疗之后从对象中获得的癌细胞的后续状态确定。在之前获得的癌细胞中确定的癌症状态、在施用治疗之后获得的癌细胞中确定的癌症状态可指示治疗对对象中癌症的效力。例如,在从治疗ecdna与癌基因a之间的相互作用之后但无ecdna与癌基因b或癌基因c之间的相互作用的适应证的对象中获得的癌细胞中的发现支持了癌症治疗在对象中的效力并且可以确定治疗降低了ecdna与癌基因b和癌基因c之间的增强相互作用的效力。
[0065]
通过ecdna激活的癌基因的一些非限制性实例是:表皮生长因子受体(epidermal growth factor receptor,egfr)、小鼠双微体2(mouse double minute 2,mdm2)、周期蛋白依赖性激酶4(cyclin-dependent kinase 4,cdk4)和cmyc。可用于治疗其中特定癌基因被激活的癌症的药剂包括但不限于:egfr抑制剂、mdm2抑制剂、cdk4抑制剂和cmyc抑制剂。在一个非限制性实例中,使用本发明方法确定为包含通过ecdna与egfr癌基因相互作用而使egfr扩增的癌症可通过向患有癌症的对象施用酪氨酸激酶抑制剂(tyrosine kinase inhibitor,tki)药物来治疗。tki的一些非限制性实例是:吉非替尼(gefitinib)和厄洛替尼(erlotinib)。在另一个非限制性实例中,使用本发明方法确定为包含由ecdna与cdk4癌基因相互作用这样的相互作用引起的cdk4转录提高的癌症可通过施用帕博西尼(palbociclib)和瑞博西尼(ribociclib)中的之一或二者来治疗。基于本文中提出的教导,技术人员将能够至少部分地基于受助于对导致癌基因转录提高的ecdna与癌基因之间的一种或更多种相互作用的鉴定来选择其他本领域已知的治疗。
[0066]
针对癌症的治疗的一些非限制性实例可包括向被诊断患有癌症、处于提高的患癌症风险中或被认为患有癌症的对象施用有效量的干扰和降低ecdna与该ecdna的靶癌基因之间相互作用的药剂。在细胞中的ecdna基因调节系统(grs)中,ecdna包括可被称为“基因调节子”序列的序列,其一些非限制性实例是ecdna中的基因致动子(actuator)序列和基因沉默子序列。包括ecdna的grs还包括“转录因子”,其是用作ecdna与其靶癌基因之间的“接触”的复合物。转录因子可包含通过转录因子与靶癌基因的“基因调节子元件”的结合将ecdna基因调节子与ecdna靶癌基因连接的多肽的复合物。例如但不旨在进行限制,控制靶癌基因转录的启动子。在一些实施方案中,本发明的方法包括鉴定存在于ecdna与其靶癌基因之间的相互作用中的候选靶标,其中所鉴定的候选靶标的破坏破坏了ecdna与其靶癌基因之间的相互作用并降低了ecdna对其靶癌基因的转录增强。作为一个非限制性实例,可将转录因子复合物中的多肽确定为候选靶标,其在与破坏grs的治疗剂接触时降低ecdna对靶
癌基因的转录增强。
[0067]
在本发明的一些实施方案中,所述方法包括接触癌细胞和/或向对象施用这样的治疗剂:其破坏grs中的候选靶标并降低ecdna与其靶癌基因之间的相互作用,从而降低ecdna对其靶癌基因的转录增强。在本发明的一些实施方案中,候选靶标包含ecdna上的基因致动子。本文中使用的术语“基因致动子”和“基因增强子”可在提及基因调节子时互换地使用。在本发明的一些方面中,候选靶标是转录因子的一种或更多种组分。在本发明的一些实施方案中,候选靶标是基因调节子元件,例如但不限于ecdna靶癌基因的启动子元件。因此,在本发明的某些实施方案中,ecdna激活子、转录因子和基因调节子元件中的一种或更多种可被确定为候选靶标,针对其指导一种或更多种治疗剂来治疗癌症。
[0068]
在本发明的一些实施方案中,治疗剂可与第二治疗剂组合施用。在一些实施方案中,药剂与癌症治疗剂组合或与另一癌症治疗(例如但不限于放射治疗、化学治疗、手术等中的一种或更多种)组合施用,例如,在癌症治疗剂的给药或施用之前、之后或与其穿插施用。在一些实施方案中,将本发明的药剂施用于经受常规化学治疗和/或放射治疗的对象。在一些实施方案中,癌症治疗剂是化学治疗剂。在一些实施方案中,癌症治疗剂是免疫治疗剂。在一些实施方案中,癌症治疗剂是放射治疗剂。
[0069]
细胞
[0070]
应理解,包括在本发明方法中的细胞可以是多个细胞之一。本文中使用的术语“多个”细胞可意指细胞群。多个细胞可均是相同类型和/或可均具有相同的疾病或病症。作为一个非限制性实例,细胞可从肝细胞群中获得,并且从该细胞群中获得的其他细胞也将是肝细胞。在本发明的一些实施方案中,多个细胞可以是混合的细胞群,意味着细胞不都是相同类型。在另一个非限制性实例中,细胞可以是从多种癌细胞获得的癌细胞。在本发明方法的一个实施方案中使用的细胞可以是以下中的一种或更多种:单细胞、分离的细胞、作为多个细胞之一的细胞、作为两个或更多个互相连接细胞的网络中的细胞的细胞、作为与彼此物理接触的两个或更多个细胞之一的细胞等。
[0071]
在本发明的一些方面中,细胞可从活的动物(例如哺乳动物)获得或者可以是分离的细胞。分离的细胞可以是原代细胞,例如最近从动物中分离的那些(例如,分离之后没有经历或仅经历少数群体倍增和/或传代的细胞),或者可以是能够在培养物中延长增殖(例如,超过3个月)或在培养物中无限增殖(永生化细胞)的细胞系的细胞。在本发明的一些实施方案中,细胞是体细胞。体细胞可从个体(例如人)中获得,并根据本领域普通技术人员已知的标准细胞培养方案进行培养。细胞可从手术标本、组织或细胞活检等中获得。细胞可从任何目的器官或组织中获得,包括但不限于:皮肤、肺、软骨、脑、乳房、血液、血管(例如,动脉或静脉)、脂肪、胰腺、肝、肌肉、胃肠道、心脏、膀胱、肾、尿道和前列腺。在本发明的一些实施方案中,细胞是hf-3035细胞或hf-2354细胞。
[0072]
在一些实施方案中,与本发明结合使用的细胞可以是健康的正常细胞,其未知具有疾病、障碍或异常病症。在一些实施方案中,与本发明的方法和组合物结合使用的宿主细胞是异常细胞,例如,从被诊断为患有障碍、疾病或病症的对象获得的细胞,包括但不限于:退行性细胞、患有神经系统疾病的细胞、疾病或病症的细胞模型、受损细胞等。在本发明的一些实施方案中,细胞可以是对照细胞。在本发明的一些方面中,宿主细胞可以是疾病或病症的模型细胞。
[0073]
可用于本发明某些实施方案的细胞是人细胞。可用于本发明方法的一个实施方案中的细胞的一些非限制性实例是以下中的一种或更多种:真核细胞、脊椎动物细胞,在本发明的一些实施方案中可以是哺乳动物细胞。可用于本发明方法的细胞的一个非限制性实例是:脊椎动物细胞、无脊椎动物细胞和非人灵长类细胞。另外,可用于本发明方法的一个实施方案中的细胞的一些非限制性实例是以下中的一种或更多种:啮齿动物细胞、狗细胞、猫细胞、禽类细胞、鱼类细胞、从野生动物获得的细胞、从家养动物获得的细胞和其他合适的目的细胞。在一些实施方案中,细胞是胚胎干细胞或胚胎干细胞样细胞。在一些实施方案中,细胞是神经元细胞、胶质细胞或者其他类型的中枢神经系统(central nervous system,cns)或外周神经系统(peripheral nervous system,pns)细胞。在一些实施方案中,细胞是星形胶质细胞。在本发明的一些实施方案中,细胞是天然细胞,以及在本发明的某些实施方案中,细胞是经改造细胞。
[0074]
可用于本发明方法的一些实施方案中的细胞可在其分离之后保持在细胞培养物中。在本发明的多个实施方案中,细胞可被遗传修饰或未被遗传修饰。细胞可从正常或患病组织获得。在一些实施方案中,细胞从供体获得,并且使用本发明方法对其状态或类型进行离体修饰。在本发明的某些实施方案中,细胞可以是培养物中的游离细胞,从对象获得的游离细胞,在来自对象、器官或固体培养物的固体活检中获得的细胞等。
[0075]
本发明的任何实施方案中的分离的细胞群或多个分离的细胞可主要或基本上完全由特定细胞类型或处于特定状态的细胞构成。在一些实施方案中,分离的细胞群由至少30%、40%、50%、60%、70%、80%、90%、95%、96%、97%、98%、99%或100%的特定类型或状态的细胞组成(即群体至少30%、40%、50%、60%、70%、80%、90%、95%、96%、97%、98%、99%或100%是纯的),例如,如通过一种或更多种标志物的表达或任何其他合适的方法所确定的。
[0076]
对照
[0077]
本发明方法的某些实施方案用于评估以下中一项或更多项:ecdna对靶癌基因的作用、细胞对于ecdna/癌基因相互作用的状态,候选治疗对ecdna与其靶癌基因之间相互作用的作用等。细胞、组织和/或对象中ecdna/靶癌基因特征的这样的评估可通过将从样品细胞、组织或对象中获得的结果与在对照细胞、组织或对象中获得的结果分别进行比较来进行。作为一个非限制性实例,本发明的一些实施方案包括在样品癌细胞中和在对照癌细胞中确定一种或更多种ecdna靶癌基因的状态,并将结果进行比较作为样品癌细胞和对照癌细胞状态差异的量度。在另一个非限制性实例中,在患有癌症的对象中鉴定ecdna/靶癌基因相互作用的状态,随后向对象施用旨在破坏所鉴定的ecdna/靶癌基因相互作用的候选治疗剂,并对施用候选治疗剂之前和之后的状态进行比较。应理解,从尚未与候选治疗剂接触的对象获得的结果可被称为“对照结果”,而未经接触的对象称为“对照对象”。
[0078]
本文中使用的对照可以是如上所述的并且也可以是可采取多种形式的预定值。其可以是单个截止值,例如中位数或平均值。其可基于比较组来建立。比较组的另一些实例可包括具有特定癌症或ecdna/靶癌基因状态的细胞或对象以及无特定癌症或ecdna/靶癌基因状态的细胞或对象。另一比较组可以是来自具有癌症家族史的组的对象以及来自无这样的家族史的组的对象。可整理预定值,例如,其中基于测试结果将受试群体均等地(或不均等地)分组。本领域技术人员能够选择用于本发明比较方法中的合适的对照组和值。
[0079]
本发明的候选治疗剂鉴定方法可在对象中的或在培养中的一种或更多种细胞中或者在体外宿主细胞中进行。在对象中进行的本发明的候选治疗剂鉴定方法可包括将旨在破坏ecdna/靶癌基因相互作用的候选药剂递送到对象的细胞中,以及评估ecdna/靶癌基因相互作用和癌基因状态(在递送候选治疗剂之前和/或之后)。可测量使宿主细胞、组织和/或对象与候选治疗剂接触的结果并将其与对照值进行比较,作为对候选治疗在破坏ecdna/靶癌基因相互作用的效力的确定。
[0080]
组合物
[0081]
本发明方法中使用的组合物可以但不必是药物组合物。本文中使用的术语“药物组合物”意指包含可用于制备通常是安全的、无毒性的、且在生物学上或其他方面都不是不期望的药物组合物的至少一种可药用载体的组合物。药物组合物可用于本发明方法的某些实施方案中,其一个非限制性实例是用于向细胞或对象施用候选治疗剂以破坏ecdna/靶癌基因相互作用。
[0082]
在本发明的某些方面中,药物组合物包含一种或更多种治疗剂或候选治疗剂,以及也施用于细胞和/或对象的一种或更多种另外的分子、治疗剂、候选药剂、候选治疗和治疗方案。在本发明方法的一个实施方案中使用的药物组合物可包含有效量的进行以下中一项或更多项的候选治疗剂:降低ecdna/靶癌基因相互作用、在癌细胞中改变靶癌基因转录的状态等。在本发明的一些实施方案中,本发明的药物组合物可包含可药用载体。
[0083]
可药用载体包括稀释剂、填充剂、盐、缓冲剂、稳定剂、增溶剂和本领域公知的其他物质。示例性的可药用载体描述于美国专利no.5,211,657中并且其他可药用载体是本领域技术人员已知的。在本发明的某些实施方案中,这样的制剂可包含盐、缓冲剂、防腐剂、相容载体、水溶液、水等。
[0084]
可通过本文中所述的多种手段和其他本领域已知的手段来实现将治疗剂递送至细胞或对象。这样的施用可进行一次或多次。如果向对象施用多次,则一种或更多种治疗剂可通过单一途径或不同途径来施用。例如但不旨在进行限制:可直接进行向待治疗对象的组织中第一次(或前几次)施用,并且随后的施用可以是全身性的。
[0085]
在本发明的某些实施方案中,递送至细胞或对象的治疗剂的量可以是统计学上显著降低ecdna与其靶癌基因的相互作用的量。合适的量可由实践者使用本文中提供的教导结合本领域已知的方法(例如临床试验)来容易地确定,并且不需要过度的实验。
[0086]
实施例
[0087]
实施例1
[0088]
在细胞核内,染色体广泛折叠成染色质环,占据不同的染色质区域[zheng,s.et al.(2013)genes dev 27,1462-1472]。这样的高度组织的三维染色质构象通过将远端调节元件及其靶基因聚集成紧密的空间接近,为许多基因组功能包括转录提供了拓扑基础[cremer,t.&cremer,m.(2010)cold spring harb perspect biol 2,a003889]。由于染色体重排导致染色质构象的改变与许多人疾病,特别是与癌症相关[sexton,t.&cavalli,g.(2015)cell 160,1049-1059]。为了解ecdna的染色质组织,以及其如何促进基因转录调节,应用了染色质相互作用评估方法,例如chia-pet[taberlay,p.c.et al.(2016)genome res 26,719-731]。设计了在相同的神经球细胞系上整合一般空间染色质组织[zhang,y.et al.(2013)nature 504,306-310]和蛋白质因子介导的长程染色质相互作用二者的方法。研究
表明,已知的ecdna通过其强烈和异常的分子内和分子间全基因组染色质接触可容易地识别。另外,在破译rna聚合酶ii(rnapii)介导的ecdna连接组及其染色体配偶体时,还鉴定了ecdna与活跃表达的常染色体癌基因之间的相关性。这一关系支持了ecdna用作可移动转录增强子来促进肿瘤进展的发现。
[0089]
方法
[0090]
gbm患者肿瘤来源神经球细胞的培养
[0091]
产生神经球细胞系并进行培养,如[decarvalho,a.c.et al.(2018)nat genet 50,708-717]所述。在亨利福特医院机构审查委员会(henry ford hospital institutional review board)批准的方案下,在征得患者书面知情同意的情况下获得脑肿瘤标本。简言之,将肿瘤标本分离并在补充有n-2补充物(17502-048,gibco)和生长因子(egf和fgf-碱性)的dmem/f12培养基(11330-032,gibco)中培养为神经球。收集传代数为15至26的细胞用于实验。
[0092]
chia-pet实验和数据分析
[0093]
将一千万个细胞在室温(rt)下用1.5mm egs(21565,thermo fisher)进行双交联45分钟,随后用1%甲醛(f8775,sigma)进行双交联20分钟,并随后用0.125m glycine(g8898,sigma)淬灭10分钟。将经交联的细胞用1
×
pbs洗涤两次,并在100μl的0.55%sds中在室温、62℃和37℃下依次各孵育10分钟进行裂解,随后添加25μl 25%triton-x 100在37℃下将sds淬灭30分钟,并添加50μl alui(r0137l,neb)、50μl 10
×
cutsmart缓冲液和275μl h2o在37℃下过夜以裂解染色质。将沉淀的经消化的细胞核重悬于500μl的含有50μl 10
×
cutsmart缓冲液、10μlbsa(b9000s,neb)、10μl的10mm datp(n0440s,neb)、10μl klenow(3
’‑5’
exo-)(m0202l,neb)和420μl h2o的da-加尾溶液中,在室温下孵育1小时,并随后通过添加200μl 5
×
连接缓冲液(b6058s,neb)、6μl生物素化桥接头(200ng/μl)、10μl t4 dna连接酶(m0202l,neb)和284μl h2o进行邻近连接并在16℃下孵育过夜。然后将经连接的染色质通过声处理进行剪切并用抗rnapii抗体(920102,biolegend)进行免疫沉淀。按[tang,z.et al.(2015)cell 163,1611-1627]所述进行经免疫沉淀dna的标签片段化(tagmentation)、生物素选择、文库制备和测序。
[0094]
chia-pet utilities(chia-pet工具的可扩展再实施方式)[li,g.et al.(2010)genome biol 11,r22](参见代码可用性)用于处理chia-pet数据。在去除测序衔接子之后,鉴定了具有桥接头的双端读段,并提取了侧翼有接头的标签。使用bwa对齐[li,h.&durbin,r.(2009)bioinformatics 25,1754-1760]和mem[li,h.(2013)arxiv:1303.3997[q-bio.gn]]将所鉴定的标签(≥16bp)根据其标签长度映射至hg19。将唯一映射的非冗余配对末端标签(pet)分类为染色体间(来自不同染色体的左标签和右标签)、染色体内(基因组跨度》8kb的左标签和右标签)和自连接pet(基因组跨度《8kb的左标签和右标签)。染色体间和染色体内pet二者均延长了500bp。然后将在两端重叠的pet聚类为ipet-2、3
……
。本研究中未检查与chr m、chr y重叠的相互作用。为了消除由tn5消化产生的基因组序列背景和标签片段化偏倚引起的潜在噪声,过滤了其中锚与黑名单(blacklist)(关于其定义见下文)重叠的相互作用。对于染色体内相互作用,使用chiasigscaled(chiasig的可扩展再实施方式)[paulsen,j.et al.(2014)nucleic acids res 42,e143]进行相互作用显著性的统计学评估。限定为ipet≥3、fdr《0.05的显著相互作用和ipet≥2的染色体间相互作用用于所
有下游分析,但nstif分析除外,其使用所有报道的染色体间相互作用(参见下文关于从原位chia-pet数据发现已知ecdna区域的部分)。使用macs2(选项:
‑‑
keep-dup all
‑‑
nomodel
‑‑
extsize 250)[liu,t.(2014)methods mol biol 1150,81-95]用所有唯一映射的读段识别rnapii结合峰。为了定义ecdna内相互作用,使用ecdna区域来收集报道区域内的所有相互作用。对于ecdna介导的反式染色体相互作用,仅包括了来源于其中ecdna驻留的染色体之外的染色体的相互作用。检查相互作用的两个锚处的rnapii结合状态,并将rnapii介导的相互作用限定为与两个锚处的rnapii结合的相互作用。所述相互作用基于其与gencode基因模型(第19版,排除了所有假基因和除mirna之外的所有rna)重叠的锚进一步进行分类,其中优先考虑了启动子(p)区域(限定为
±
2.5kb的tss),随后是基因区域(g)。不与任何基因内区域重叠的锚被归类为基因间的(i)。来自ncg6联合列表[repana,d.et al.(2019)genome biol 20,1]和cosmic v87[forbes,s.a.et al.(2015)nucleic acids res 43,d805-811]]的癌基因用于注释与ecdna相互作用的基因。
[0095]
黑名单区域
[0096]
为了消除chia-pet实验程序引入的偏倚,例如tn5在基因组上某些基因座中的过度标签片段化,使用由不同抗体富集制成的8个人细胞chia-pet文库(4个用抗ctcf抗体和4个用抗rnapii抗体)来创建灰名单(greylist),将其降采样(down-sample)以表示同等数目的读段,总计75,215,727个标签。使用macs2.1.0.20151222[liu,t.(2014)methods mol biol 1150,81-95]用fdr《0.05从合并数据集中识别峰,产生了153,735个峰。常染色体和x染色体上的这些峰区域是灰名单的候选者。将具有较短峰的区域(可能是由于tn5标签片段化)进行进一步过滤,同时保持由q值测量的最高置信度,并按以下标准堆积(pileup):区域长度《600bp,前1%最高堆积,10%最低q值,以及前10%最高的峰倍数富集。获取经过滤峰(q值《1e-165且倍数富集为10至50的1,119个区域)的这3个量或向量(反向长度、倍数富集和堆积),并且缩放(scale)每个向量(用r命令“scale(center=f、scale=t)”)。接下来,将这三个向量单独归一化,使得每个向量的平均值为1。定义了评分函数,s=倍数富集+堆积+1/长度。灰名单由评分s高于平均值的321个峰区域组成。另外,包括了显示是来自目视检查的伪影的四个区域。采用的最终黑名单是chia-pet灰名单和来自kundaje实验室的可公开得到的黑名单(github.com/kundajelab/hic-pipeline/blob/master/hic_flexiblewindow-pipel ine/data/reference_genomes/hg19/wgencodehg19consensussignalartifactre gions.bed.gz)的结合(concatenation)。
[0097]
从原位chia-pet数据中发现已知ecdna区域
[0098]
将来自各细胞系的rnapii chia-pet的经处理的相互作用数据聚集到全基因组相互作用频率(interaction frequency,if)矩阵m
nxn
={m
ij
|i,j=1,2,..,n}中。将hg19基因组从染色体起始以50kb间隔区段化成60,739个不重叠区块(每个染色体的最后一个区块可能不代表完整的50kb)。从if矩阵中去除了与黑名单(见下文)重叠的区块。已知的ecdna区域在ecdna区域内和在遍布所有23对染色体中都表现出大数目的相互作用,产生了非常高的if总和,特别是在ecdna上扩增的不同染色体区域之间。所使用的方法利用这些特征来测试已知ecdna区域[decarvalho,a.c.et al.(2018)nat genet 50,708-717]是否可从chia-pet相互作用数据中发现,并可预测在ecdna上扩增的另外的基因组区域。
[0099]
计算每个区块的反式染色体if(tif)总和并将其归一化,使得其在不同文库中是
相当的。将该数据向量缩放(除以其幅度并乘以向量的长度)使得平均值等于1。使该归一化向量的第i个区块为nstifi。将具有最高nstif的区块与ecdna(+)数据中的已知ecdna区域进行比较。为了解nstif在来自ecdna(-)细胞的chia-pet数据中的分布,在hf-3035细胞(图1b)以及其他多能细胞系(数据未示出)中检查了nstif的全基因组分布并发现它们均小于20。因此,引入阈值作为确定ecdna候选区域的第一遍(first pass)。还引入了nstif的另外的阈值,以将区块优先作为ecdna的候选者。基于知识,由ecdna扩增的基因组区域不超过多于0.1%的基因组大小,因此将低阈值(t
l
)设置为前0.1%中nstifi的平均值。根据经验,如果t
l
《25,则将高阈值(th)设置为25(即高于所预计的25倍),否则th=最大(nstif)。具有nstifi》t
l
的所有第i个区块都被放入候选者的列表中。在另一方面,如果所有nstifi《th,则没有区域将被视为ecdna。在本研究中,来自ecdna(-)数据的最高nstif低于21,这显著低于ecdna(+)数据集中的那些(约38至82)。
[0100]
将候选者列表基于其基因组距离进行分组,使得位于彼此附近的组被分组在一起,即2个组之间的最小距离为1mb。分组的目的是根据与不同组中候选者的承诺连接(committed connection)(由if测量)对每个候选者进行评分。该评分是基于每个候选者所连接多少组。如果候选者的归一化tif高于阈值t
t
,则定义为“连接”。使矩阵m的归一化染色体间元素为t
ij
,并且区块i的归一化tif向量为ti(与nstif的归一化相同,即(与nstif的归一化相同,即其中j是索引i的反式染色体})。如果连接相互作用频率相对较高,则存在连接,例如存在至少一个区块,例如第k个区块,在另一组候选者中t
ik
》t
t
,其中t
t
是100的较高值和前0.1%ti的平均值。然后将连接评分+1添加至候选i。
[0101]
然后首先检查具有最高nstif的候选组,以查看其连接评分中的全部是否都为零。当此条件为真时,如果不超过5个组nstif≥th,则建议该单一组中的所有候选区域都是ecdna区域,否则无法预测ecdna区域(噪声数据可显示出许多组显示出高于高阈值的nstif且连接评分为0,这可能是假阳性)。当条件为假时,即存在具有非零连接评分的多个组,预测来自连接评分大于零的那些组的区域是ecdna。进行了另外的研究,以分配在来自chia-pet数据的ecdna上扩增的区域的特异性。
[0102]
chip-seq文库构建和数据分析
[0103]
以与chia-pet相同的方式对200万个细胞进行交联和裂解。在裂解之后,对细胞核沉淀进行声处理并用抗h3k27ac抗体(39133,active motif)进行免疫沉淀。用kapa hyper prep试剂盒(kk8505,kapa biosystems)对来自抗体免疫沉淀和输入的4ng dna进行端修复、a加尾和衔接子连接。将经衔接子连接的dna片段用kapa library amplification ready mix(kk2612,kapa biosystems)进行pcr扩增,并在illumina平台上用75bp单端测序进行测序。将原始读段使用trim galore 0.4.3版(选项:
‑‑
stringency 3-q 30-e.20
–
length 15)进行品质修整,并使用bwa 0.7.12(命令:aln)[li,h.&durbin,r.(2009)bioinformatics 25,1754-1760]映射到hg19基因组。使用唯一映射和去重复的读段用macs2.1.0.20151222(选项:
‑‑
nomodel
‑‑
extsize 250-b
‑‑
spmr-g hs)[liu,t.(2014)methods mol biol 1150,81-95]进行峰识别(peak calling)(fdr《0.05)。在所有分析中使用限定为fdr《0.05的峰。对于图2c至d中的h3k27ac分析,还应用了p《0.001的另外的要求。
[0104]
免疫染色
[0105]
将未固定的中期细胞滴至载片上并将其在kcm缓冲液(120mm kcl、20mm nacl、10mm tris-hcl、ph 8.0、0.5mm edta、0.1%(v/v)triton x-100)中在室温下预孵育10分钟。将载片在1%(w/v)bsa/kcm缓冲液中在室温下封闭30分钟,随后在2%bsa中与第一h3k27ac抗体(39133,active motif)一起在4℃下孵育过夜,与kcm缓冲液在室温下孵育10分钟,进行两次,与山羊抗兔alexa fluor 488二抗(a32731,invitrogen)在室温下孵育30分钟。在用kcm缓冲液洗涤两次之后,将载片在4%(v/v)甲醛/kcm中交联15分钟,用50μl prolong gold antifade(invitrogen)封固盖片并用透明甲油密封。在leica sted 3x/dls confocal下扫描载片。
[0106]
转录因子基序分析
[0107]
homer2[heinz,s.et al.(2010)mol cell 38,576-589]针对206个靶序列中的414个已知基序在71,232个h3k27ac峰区域的归一化背景下进行搜索。基于以下标准选择搜索结果:q值《0.001,富集》1.5,具有基序的靶序列的百分比》25%。
[0108]
rna-seq文库构建与数据分析
[0109]
使用allprep dna/rna mini kit(80204,qiagen)以生物学重复分离总rna。使用kapa stranded mrna sequencing kit(kk8502,kapa biosystems)按照制造商说明从300ng总rna生成链特异性rna文库。将文库在illumina平台上用75bp双端测序进行测序。将原始测序读段使用trim galore 0.4.3版(选项:
‑‑
stringency 3-q 20-e.20
‑‑
length 15
‑‑
paired)进行修整,并使用hisat 2.1.0(选项:
‑‑
dta-cufflinks)与hg19基因组对齐。使用cufflinks 2.2.1[trapnell,c.et al.(2010)nat biotech 28,511-515]对转录物进行组装,并使用cuffdiff(选项:
‑‑
library-type fr-firststrand)[trapnell,c.et al.(2013)nat biotech 31,46-53]对最终表达水平进行定量。用r/pheatmap包(1.0.2.版)分析来自不同样品的测序数据之间的相关性。限定ecdna介导的染色质相互作用群落
[0110]
对由rnapii结合在两个锚上支持的所有反式染色体相互作用进行收集。将来自hf-2354、hf-2927、hf-3016和hf-3177细胞系的所有rnapii结合锚合并,并且将与黑名单区域重叠的锚去除以合并到29,721个非重叠相互作用节点中(图3c)。对于每个节点,限定了连接评分,即其所连接的相互作用配偶体节点的数目。将枢纽限定为具有高于大于平均值3倍标准偏差的连接评分的节点。在该参数的情况下,枢纽具有》10个与其他节点连接的连接。总共从hf-2354、hf-2927、hf-3016和hf-3177细胞系中分别限定了69、106、82和99个枢纽。使用枢纽-枢纽网络,在r的igraph文库[csardi,g.&nepusz,t.(2006)computer science]中用cluster_edge_betweenness函数生成群落。来自hf-2354、hf-2927、hf-3016和hf-3177细胞系的ecdna相关群落的数目分别为8、8、10和4个。
[0111]
数据可用性声明
[0112]
本研究中所述的所有数据均储存在ncbi的gene expression omnibus gse124769中,使用以下链接www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=gse124769用审阅连接密钥“uxkhaey ctdixts”。
[0113]
代码可用性
[0114]
从chia-pet数据中进行ecdna检测的代码可在www.dropbox.com/sh/2crbfjt1kr2yyws/aacliug6ch9y6furbbuzmcbpa?dl=0上获得。chia-pet工具(代码可在github.com/cheehongsg/cpu上获得)chiasigscaled(代码可在github.com/cheehongsg/
chiasigscaled上获得)。
[0115]
结果和讨论
[0116]
对五种gbm患者来源神经球细胞系进行了chia-pet分析,这些细胞系的ecdna状态先前从全基因组测序数据中建立,并通过荧光原位杂交(fluorescence in-situ hybridization,fish)分析进行了确定。五种神经球系中有四种是ecdna(+)(hf-2354、hf-2927、hf-3016和hf-3177),而一种系是ecdna(-)(hf-3035)[decarvalho,a.c.et al.(2018)nat genet50,708-717]。基于从ecdna内扩增的基因表达的高水平rna(图4a),推断出ecdna与活性染色质结构域内的rnapii高度关联。rnapii染色质免疫沉淀用于牵出rnapii相关染色质,并使用chia-pet测定来表征ecdna-染色质相互作用组(图5a)。所得chia-pet数据检出rnapii结合位点、调节元件之间的长程染色质相互作用[zhang,y.et al.(2013)nature 504,306-310](图4b)以及空间拓扑染色质相关结构域(tad)内的非富集染色质接触[dixon,j.r.et al.(2012)nature 485,376-380](图6a)。染色体结构变异,例如染色体10q23和9p21上pten以及cdkn2a和cdkn2b的缺失,导致染色质接触的消除(图6b)。另一些实例包括检出hf-2927中涉及dmd基因的chrx:31.4-32mb常见脆弱位点的600kb缺失[ma,k.et al.(2012)int j mol sci 13,11974-11999],hf-2354基因组中chr3:168-183mb的15mb广泛重排以及chr3与chr3之间3.5和11.5mb的双易位事件(图6c)。
[0117]
hf-2927具有chr7p11/含ecdna的egfr[decarvalho,a.c.et al.(2018)nat genet 50,708-717],被称为ecegfr,而hf-2354包含chr8q24/myc ecdna,被称为ecmyc。在hf-3016和hf-3177(来源于同一患者的原发性和复发性gbm的两种神经球系)中,发现三个基因在染色体外进行扩增[decarvalho,a.c.et al.(2018)nat genet 50,708-717],其中表明chr7p11/egfr和ch12q14.1/cdk4在ecdna上共扩增,而chr8q24/myc也在ecdna上发现(分别被称为ecegfr、eccdk4和ecmyc)。所有ecdna基因座在整个基因组中都表现出广泛的接触(图5b,图1a),这表明这些ecdna区域与所有染色体中的区域均具有高反式连接性。为了量化反式染色质接触的程度,开发了如实地描述在所有23对染色体中归一化的全基因组反式相互作用频率(nstif)的度量,并将该度量应用于五种神经球细胞系中的每一个(参见本文中的方法)。ecdna区域在所有四种ecdna (+)系中都显示出高度升高的nstif水平(最大nstif为约38至82),但在hf-3035ecdna(-)细胞(nstif《21)中没有(图5b,图1b)。nstif尖区域(spike region)与ecdna区域紧密匹配。此外,与ecdna区段连接的高nstif区域显示出整个基因组中的反式接触(图5b),表明来自染色体外遗传元件的动态ecdna连接性。特征性的全基因组接触模式可用ecdna的可移动性质来解释。在两种双扩增子系hf-3016和hf-3177中,结果显示出ecmyc、ecegfr和eccdk4 nstif水平升高(图1b)以及三个基因座之间的交叉相互作用,表明这些系中的显性ecdna携带所有三个癌基因或其具有紧密的分子间接近度。为了验证高nstif对ecdna的染色体外性质而非扩增状态具有特异性,将来自具有拷贝数增加≥3(其为3至6)的所有基因组区域的nstif值与来自ecdna区域的nstif进行比较。来自如预期被限制在染色体区域内的染色体扩增区段的nstif值显著低于(中位数nstif为1.5至4vs.24至43)来自ecdna区域的nstif(单侧wilcoxon秩和检验,p值《0.0005)(图5c),确定了ecdna在整个基因组中接触频率的升高不仅仅由dna剂量效应来解释,而是由其自主能力所决定。
[0118]
除了高反式相互作用频率之外,在ecdna的基因组区域内还检出了显著高频率的
顺式相互作用。在hf-2927ecdna(+)细胞中,在约530kb ecdna区域内观察到的顺式相互作用强度为2,879,与在ecdna(-)hf-3035细胞中的相同区域仅为12相比提高了240倍。高接触提高直接反映了该ecegfr的尺寸和基因组结构二者。类似地,在hf-2354中限定的ecmyc区域的两个区段内和两个区段之间观察到强烈的顺式相互作用(图7a)。广泛的rnapii系留的染色质接触(定义为在通过相互作用连接的dna区域检出的rnapii结合,被称为锚)以ecdna内不同区域之间的顺式作用(被称为ecdna内)(图7b,图8a)以及与线性染色体上的其他基因或调节元件的反式作用(被称为反式相互作用)(图7c)检出。染色体外内连接模式显示出具有高频率相互作用和强烈接触焦点的显著环对(图7b,图8a),其可共同来源于不同ecdna分子之间的接触和单独ecdna内的折叠。这一假设通过原发性和匹配的复发性神经球系(hf-3016vs.hf-3177)得到了证实,虽然这两种系都包含ecmyc、ecegfr和eccdk4,但在hf-3016和hf-3177中检出的92%和95%的ecdna内环仅在其各自的细胞中发现。在hf-3016中检出的ecdna内环主要位于eccdk4基因座中(图8a中的内圈),而在hf-3177中,ecmyc区域显示出更紧密的成环(图7b中的内圈),这可暗示ecdna虽然可涉及相似的癌基因驱动子,但来自不同和独特的结构。在ecdna与其染色体配偶体之间的反式相互作用中,ecdna上的锚主要(75至93%)在基因内或基因间非编码区域,而其反式相互作用染色体锚主要(79至84%)位于启动子(限定为tss
±
2.5kb)处(图7c)。这样的相互作用并置表明了这些接触的转录功能。
[0119]
为了解决ecdna相互作用如何与转录调节区域相关联的问题,进行了h3k27ac谱分析以标记活性增强子和启动子。首先检查了在ecdna上扩增的癌基因的调节,通过评价其反式染色体相互作用区域来进行。这些ecdna连接非编码染色体锚表现出与h3k27ac峰高重叠(基因间61至80%);其显著高于来自无ecdna接触的反式相互作用非编码染色体锚的那些(基因间38至69%,图8b,p值0.019,单侧wilcoxon秩和检验)。具体而言,与驻留于ecdna上的癌基因的启动子相互作用的73%(196个中144个)染色体非编码区域与其相应细胞系中的h3k27ac峰重叠(图2a),表明通过染色质接触与线性染色体上的增强子接合进一步增强了ecdna上癌基因的转录。增强子接触是可变的,并且在不同的ecdna(+)系中可以是动态的。在ecmyc的情况下,在三种ecmyc(+)细胞系中,myc启动子与九个不同的h3k27ac增强子相互作用(图8c)。
[0120]
观察到在ecdna内在高频率接触焦点与h3k27ac峰之间的共同出现(图7b,图8a),表明这些相互作用锚表现类似于活性增强子。530kb ecegfr区域内的h3k27ac峰与hf-2927中的高相互作用频率区域共对齐,并且当与ecdna(-)细胞(hf-3035)中染色体egfr区域中的h3k27ac峰相比时,表现出作为在更广泛的基因组跨度下密切接近的簇的模式,表明增强子信号在ecdna的染色质接触位点上积累。使用靶向h3k27ac的抗体针对中期hf2927细胞进行的免疫染色表现出h3k27ac与标记ecdna的dapi信号之间的重叠信号,这确定了增强子功能与ecdna之间的相关性(图8d)。
[0121]
为了量化地表明与ecdna介导的反式染色质相互作用相关的h3k27ac信号在所有四种ecdna(+)细胞系中的提高,对在四种ecdna(+)细胞系中的每一种中在ecdna区域(被称为a组)、其相应的反式相互作用染色体配偶体(b组)以及无ecdna接触的全基因组h3k27ac峰(c组)之间检出的所有h3k27ac峰(fdr《0.05,p《0.001)的倍数富集进行了比较。与ecdna染色质相互作用锚相关的h3k27ac峰(中位数值:a组为58至138,且b组为43至91)与无ecdna
接触的全基因组h3k27ac峰的那些相比具有显著更高的富集(中位数值10至12,p值5e-09至2.3e-164,单侧wilcoxon秩和检验)(图2c)。其也高于在ecdna(-)hf-3035细胞系中发现的ecmyc、ecegfr和eccdk4等同区域的富集倍数(中位数值:9至11),确定了h3k27ac信号的强烈增强是ecdna所特有的。
[0122]
具有超高强度和大结构域的h3k27ac信号的增强子被称为“超级增强子”[whyte,w.a.et al.(2013)cell 153,307-319],已发现其促进癌症中的癌基因转录[hnisz,d.et al.(2013)cell 155,934-947]。为了评价在ecdna中观察到的增强子特征是否让人联想到“超级增强子”,对在ecdna及其反式相互作用染色体锚中检出的h3k27ac峰的跨度大小进行了检查。发现ecdna上的h3k27ac峰与无ecdna接触的染色体h3k27峰相比具有显著更长的跨度(中位跨度:a组中为2至3.5kb,b组中1.5至2.1kb vs.c组中700至800bp,p值9.6e-08至4.4e-153,单侧wilcoxon秩和检验)(图2d)。a组ecdna上的h3k27ac峰相对于其他h3k27ac峰区域的序列分析显示对调节rnapii一般转录和细胞增殖至关重要的转录因子(包括jun、fos和atf)结合基序的富集(q值《0.001,富集》1.5)。总的来说,rnapii信号的顺式和反式会聚与h3k27ac信号的强烈增强表明ecdna分子能够广泛地连接整个基因组的rna聚合酶机构,证实了作为全基因组转录放大器的功能。
[0123]
接下来,通过分析来自相同四种系的rna表达,确定与ecdna反式相互作用相关的增强子信号的提高是否导致活性转录。共计地,结果表明在hf-2354、hf-2927、hf-3016和hf-3177ecdna(+)细胞系中,分别检出其启动子与ecdna进行接触的1,887、1,270、1,483和1,157个染色体基因。与无其他反式染色体接触的基因(fpkm中位数值0.7至4,p值4.3e-34至1.8e-55,单侧wilcoxon秩和检验)(图9a)或与ecdna无接触但与其他基因具有反式染色体相互作用的基因(fpkm中位数值8至9,p值6.1e-04至1.5e-08,单侧wilcoxon秩和检验)(图10)相比,ecdna相互作用基因显示出显著更高的表达水平(fpkm中位数值12至14)。此外,ecdna连接基因的表达水平与其ecdna接触的频率(通过独立的反式相互作用的数目来测量)呈正相关(图9b)。总的来说,ecdna连接与转录活性和高度增强的h3k27ac特征高度相关,表明ecdna可充当全局转录扩增机构。
[0124]
在总共四种ecdna(+)细胞系中,4,763个基因与ecdna接触,其中877个(18%)由两种或更多种细胞系共有(图3a)。这些877个基因的功能在参与细胞通讯和增殖的生物学过程:翻译起始(fdr 2.06e-04)、细胞间黏附(fdr 0.003)和转录(fdr 0.02)中显著富集(david在线分析[huang da,w.et al.(2009)nat protoc 4,44-57])。在所有四种ecdna(+)细胞系中常见的20个基因中,超过一半是与肿瘤发生、凋亡调节、细胞生长或增殖功能上相关的基因,包括erbb2、dnajb4、mcl1、ddit4和bad、jund及其转录辅因子fos,以及非编码rna基因malat1。对一组736个染色体癌基因[forbes,s.a.et al.(2015)nucleic acids res 43,d805-811;repana,d.et al.(2019)genome biol 20,1]在ecdna连接网络中的存在进行了检查,并且发现在四种ecdna (+)细胞系中,分别有87、56、78和54个带注释的癌基因在ecdna介导的染色质相互作用组内(图3b)。这代表了比随机预期高2.1至2.5倍的富集(p《0.05;单侧wilcoxon秩和检验),支持了ecdna通过染色质相互作用募集另外的癌基因以在癌细胞中共激活的假设。与ecdna对转录激活的作用一致,与4种ecdna(+)系中每一种的所有基因的中位转录水平相比,反式相互作用的癌基因显示出6至10倍的fpkm提高(p值1.3e-14至2.1e-24,单侧wilcoxon秩和检验)(图9c)。已确定736个带注释的癌基因中总共216个
在四种ecdna(+)细胞系中的至少一种中表现出与ecdna的反式染色体相互作用(图3b)。其中,erbb2和malat1在所有四种神经球系中均表现出与ecdna的染色质连接。erbb2是乳腺癌中的典型癌基因,并与egfr共有结构相似性,其在55%至60%的胶质母细胞瘤中发生改变。erbb2和egfr可形成异二聚体以激活其下游信号传导途径[qian,x.,et al.(1994)proc natl acad sci u s a 91,1500-1504]。erbb2在gbm中很少发生基因组改变,并且在近一半的gbm中表达[zhang,c.et al.(2016)j natl cancer inst108,doi:10.1093/jnci/djv375;liu,g.et al.(2004)cancer res 64,4980-4986],但在非肿瘤性脑细胞中不表达(gepia在线数据[tang,z.et al.(2017)nucleic acids res 45,w98-w102])。gbm中的malat1表达可导致wnt信号传导[vassallo,i.et al.(2016)oncogene 35,12-21],其驱动内皮转分化并且提高迁移潜力[hu,b.et al.(2016)cell 167,1281-1295]。
[0125]
除了募集单独癌基因之外,ecdna还显示出是其中许多癌基因通过其与ecdna的相互作用而聚集在一起呈空间接近的焦点。在由枢纽之间的广泛通信限定的8、11、10和4个全基因组相互作用网络中,在四种ecdna(+)细胞系中的每一种中(方法,图3c),除了hf-2927中的3个,所有都具有来自ecdna的相互作用枢纽。这些ecdna连接癌基因中的许多驻留于每个染色质网络中。具体而言,hf-3016和hf-3177中的单独群落可连接多至10个和12个另外的癌基因(图9d),这显著高于随机预期(p《0.05;单侧wilcoxon秩和检验)。表明了癌基因的这样的共聚集是ecdna采用的基于结构的机制,以实现协调的转录共激活来促进肿瘤发生。值得注意的是,尽管来自hf-3016和hf-3177的ecdna来源于同一患者的原发性和复发性gbm,但ecdna网络的组合一致性并未包含许多重叠基因,这意味着癌症克隆的异质性可通过ecdna-染色质网络进一步扩大。
[0126]
总之,使用染色质相互作用测定chia-pet测定进行了研究,以在癌细胞中表征ecdna转录相互作用组和调节。通过对全基因组ecdna连接焦点、反式相互作用的染色体靶基因、h3k27ac结合和rna表达的多组学综合分析,表明了ecdna可作为可移动增强子元件发挥作用,其可在癌细胞中优先靶向癌基因以进行转录共激活(图9e)。这些发现概述了一种新的ecdna机制,其为癌细胞提供了驱动肿瘤进展和肿瘤进化的竞争优势。此外,ecdna靶向癌基因的鉴定可揭示靶向抑制治疗策略的候选者,并且由ecdna转录激活的癌基因簇可以是优先考虑其对给定肿瘤类型的有效性的机制。
[0127]
除了提供对癌症基因组中ecdna靶向染色质相互作用组的详细表征之外,染色质相互作用测定,例如但不限于chia-pet测定的使用提供了有效的手段来精确定位ecdna内扩增的基因组结构域,这是基于其与线性染色体的加强染色质接触进行的。表征ecdna所采用的现有方法是通过基于成像的分析[turner,k.m.et al.(2017)nature 543,122-125]或具有拷贝数增加的区域的结构分析[deshpande,v.et al.(2018)biorxiv doi.org/10.1101/457333]。与这些方法相比,通过染色质相互作用测定(其一些非限制性实例是chia-pet和hi-c)直接测量染色体间染色质接触频率提供了无偏方法来揭示不同尺寸、拷贝数或序列背景的ecdna特征。此外,ecdna分子不同区域之间的接触频率和模式提供了对其物理结构和连续性的见解,就像3d染色质构象有助于表征基因组结构变异和组装的能力[spielmann,m.et al.(2018)nat rev genet 19,453-467;dixon,j.r.etal.(2018)nat genet 50,1388-1398]。
[0128]
综上所述,实验结果表明,ecdna可通过染色质相互作用来增强染色体外和染色体
基因转录的表达。该发现结合ecdna的普遍性和多样性,提供了癌症中ecdna作用的另一个复杂水平。重要的是,结果提供了对在肿瘤进化中遗传结构与表观遗传学结果之间相互作用的见解。鉴于ecdna在癌症中的普遍性以及这种染色体外结构的独特基因组动力学,这支持了在治疗中靶向ecdna及其激活的染色体基因。
[0129]
等同方案
[0130]
尽管本文中已经描述和举例说明了本发明的数个实施方案,但是本领域普通技术人员将容易地预想用于进行本文中所述功能和/或获得本文中所述的结果和/或一个或更多个优点的多种其他手段和/或结构,并且这样的变化方案和/或修改方案中的每一个都被认为在本发明的范围内。更一般地,本领域技术人员将容易地理解,本文中所述的所有参数、尺寸、材料和构造均意指是示例性的,并且实际参数、尺寸、材料和/或构造将取决于使用本发明的教导的一项或更多项特定应用。本领域技术人员将认识到或者能够仅使用常规实验来确定本文中所述发明的具体实施方案的许多等同方案。因此,应当理解,前述实施方案仅通过实例给出,并且在所附权利要求书及其等同文件的范围内,本发明可以以不同于具体描述和要求保护的其他方式实施。本发明涉及本文中所述的每个单独特征、系统、制品、材料和/或方法。另外,如果这样的特征、系统、制品、材料和/或方法没有相互不一致,则两个或更多个这样的特征、系统、制品、材料和/或方法的任意组合包括在本发明的范围内。
[0131]
当提供值范围时,应理解涵盖每个中间值。当所规定范围包含一个或两个限制时,排除那些被包含的限制之一或二者的范围也包含在本发明中。
[0132]
本文中定义和使用的所有定义应理解为优先于字典定义、通过引用并入的文件中的定义和/或所定义术语的一般含义。
[0133]
除非明确相反地指出,否则如本文在说明书和权利要求书中使用的没有数量词修饰的名词应理解成意指“至少一者”。如本文在说明书和权利要求书中使用的短语“和/或”应理解成意指如此连接的要素中的“之一或二者”,即在一些情况下共同存在而在其他情况下分开存在的要素。除非明确相反地指出,否则可以任选地存在除通过“和/或”子句具体标识的要素之外的其他要素,无论其与具体标识的那些要素相关或不相关。
[0134]
本技术中引用或提及的所有参考文献、专利和专利申请以及出版物均通过引用整体并入本文。
