1.本发明属于生物医药领域,具体涉及用于卵巢癌诊断或预后风险评估的标志物。
背景技术:
2.卵巢癌(ovarian cancer,ov)是女性最常见的妇科癌症的第三大原因,也是全球女性癌症死亡的第五大原因。由于早期非特异性症状,大多数卵巢癌妇女被诊断为晚期,5年生存率为25%。
3.卵巢癌的特点是发病率较高,死亡率高,预后差。肿瘤分化差、疾病分期高、减细胞手术后残留疾病、年龄大、吸烟、体重超标、缺乏体育活动均与卵巢癌预后不良相关。虽然大多数患者最初对化疗反应良好,但部分患者出现复发并产生化疗耐药。因此,确定可改善卵巢癌患者预后的潜在预测因子具有重要的临床意义。
技术实现要素:
4.本发明的第一目的在于提供一种用于卵巢癌的预后风险模型。
5.本发明的第二目的在于提供用于诊断卵巢癌的标志物。
6.为实现上述目的,本发明采用了如下技术方案:
7.本发明第一方面提供了用于诊断卵巢癌或评估卵巢癌预后风险的标志物,所述的标志物包括akap12、aldoc、angptl4、cited2、ppp1r15a和或prdx5。
8.进一步,所述的标志物还包括isg20和/或tgfbi。
9.进一步,所述的标志物为akap12、aldoc、angptl4、cited2、isg20、ppp1r15a、prdx5和tgfbi的组合。
10.本发明第二方面提供了一种试剂,所述试剂可以检测样本中本发明第一方面所述的标志物的表达水平。
11.进一步,所述的试剂包括能够检测所述标志物的mrna的表达水平的试剂。
12.可选的,所述的检测标志物的mrna的表达水平使用选自聚合酶链反应、实时荧光定量逆转录多聚酶链反应、逆转录聚合酶链反应、竞争性聚合酶链反应、核酸酶保护分析、原位杂交法、核酸微阵列、rna印迹或dna芯片的方法中的任何一种来进行。
13.进一步,所述的试剂包括能够检测所述标志物的编码蛋白的表达水平的试剂。
14.可选的,所述的检测标志物的编码蛋白的表达水平使用选自多重邻位延伸分析、酶联免疫吸附、放射免疫测定、夹心分析、蛋白质印迹、免疫沉淀、免疫组织化学染色、荧光免疫测定、酶底物显色、抗原抗体聚集、荧光激活细胞分选、质谱分析、mrm测定、采用一组多重胺特异性稳定同位素试剂的测定或蛋白质芯片测量中的任何一种来进行。
15.进一步,所述的试剂包括:
16.与所述标志物基因特异性结合的引物或探针;
17.与所述标志物蛋白特异性结合的抗体、肽、适配体或化合物。
18.本发明第三方面提供了一种卵巢癌预后风险评估模型,所述的模型以本发明第一
方面所述的标志物表达水平为输入变量。
19.进一步,所述的模型使用以下方程计算风险得分:
20.风险得分=(0.045
×
akap12表达水平)+(0.099
×
aldoc表达水平)+(0.109
×
angptl4表达水平)+(0.096
×
cited2表达水平)-(0.306
×
isg20表达水平)+(0.046
×
ppp1r15a表达水平)-(0.169
×
prdx5表达水平)+(0.045
×
tgfbi表达水平)
21.本发明第四方面提供了一种卵巢癌预后风险评估系统,包括:
22.1)检测单元:包括本发明第一方面所述标志物的检测模块;
23.2)分析单元:将检测单元检测得到的标志物的表达水平作为输入变量,输入本发明第三方面所述的模型进行分析;
24.3)评估单元:判断样本对应的受试者卵巢癌预后的风险高低。
25.本发明第五方面提供了如下任一项所述的应用:
26.(1)本发明第二方面所述的试剂在制备用于诊断卵巢癌的产品中的应用;
27.(2)本发明第二方面所述的试剂在制备用于评估卵巢癌预后风险的产品的产品中的应用。
28.(3)本发明第一方面所述的标志物在构建本发明第三方面所述的模型中的应用;
29.(4)本发明第一方面所述的标志物在构建本发明第四方面所述的系统中的应用。
30.进一步,所述的产品包括试剂盒、芯片。
31.本发明第六方面提供了一种计算机可读存储介质,所述的计算机可读存储介质包括存储的计算机程序,其中,在所述的计算机程序运行时控制所述的计算机可读存储介质所在装置执行本发明第三方面所述的模型。
附图说明
32.图1是缺氧基因的单因素cox分析结果森林图;
33.图2是lasso cox分析结果,其中,2a是lasso回归模型系数图,2b是交叉验证结果图;
34.图3是tcga数据集中缺氧基因的的风险评分、os状态及热图,其中,3a是缺氧基因的的风险评分图,3b是os状态统计图,3c是基因表达热图;
35.图4是geo数据集中缺氧基因的的风险评分、os状态及热图,其中,4a是缺氧基因的的风险评分图,4b是os状态统计图,4c是基因表达热图;
36.图5是kaplan-meier生存分析结果图,其中,5a是tcga数据集中高低风险评分患者的生存情况统计图,5b是geo数据集中高低风险评分患者的生存情况统计图;
37.图6是评估本发明所涉及基因在生存率中的预测效率的roc曲线图,其中,6a是在tcga数据集中的时间依赖性roc曲线,6b是在geo数据集中的时间依赖性roc曲线;
38.图7是单因素cox分析结果图,其中,7a是在tcga数据集中将单因素cox分析缺氧风险信号用于评估ov患者os的独立预后价值的实验结果图,7b是在geo数据集中将单因素cox分析缺氧风险信号用于评估ov患者os的独立预后价值的实验结果图;
39.图8是多因素cox分析结果图,其中,8a是在tcga数据集中将多因素cox分析缺氧风险信号用于评估ov患者os的独立预后价值的实验结果图,8b是在geo数据集中将多因素cox分析缺氧风险信号用于评估ov患者os的独立预后价值的实验结果图;
40.图9是pcr产物电泳图;
41.图10是内参gapdh基因实时扩增曲线图及产物溶解曲线图,其中,10a是基因实时扩增曲线图,10b是产物溶解曲线图;
42.图11是内参actb基因实时扩增曲线图及产物溶解曲线图,其中,11a是基因实时扩增曲线图,11b是产物溶解曲线图;
43.图12是akap12基因实时扩增曲线图及产物溶解曲线图,其中,12a是基因实时扩增曲线图,12b是产物溶解曲线图;
44.图13是aldoc基因实时扩增曲线图及产物溶解曲线图,其中,13a是基因实时扩增曲线图,13b是产物溶解曲线图;
45.图14是angptl4基因实时扩增曲线图及产物溶解曲线图,其中,14a是基因实时扩增曲线图,14b是产物溶解曲线图;
46.图15是cited2基因实时扩增曲线图及产物溶解曲线图,其中,15a是基因实时扩增曲线图,15b是产物溶解曲线图;
47.图16是ppp1r15a基因实时扩增曲线图及产物溶解曲线图,其中,16a是基因实时扩增曲线图,16b是产物溶解曲线图;
48.图17是prdx5基因实时扩增曲线图及产物溶解曲线图,其中,17a是基因实时扩增曲线图,17b是产物溶解曲线图;
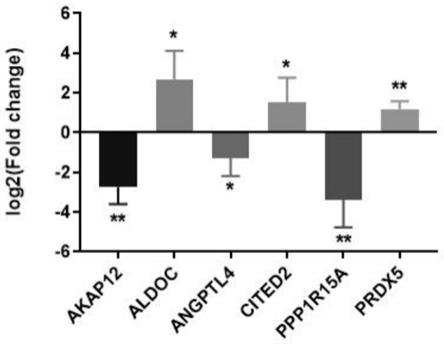
49.图18是本发明所涉及基因差异表达结果图。
具体实施方式
50.标志物
51.本发明提供了用于诊断卵巢癌或评估卵巢癌预后风险的标志物。
52.在本发明中,术语“标志物”意指化合物,优选是基因,与来自具有第二表型(例如没有疾病)的受试者或一组受试者的生物样品相比,它在来自具有第一表型(例如患有疾病)的受试者或一组受试者的生物样品中差异地存在(即增加或减少)。术语“标志物”通常是指一种基因的存在/浓度/含量或两种或更多种基因的存在/浓度/含量。
53.在本发明中,所述标志物包括akap12、aldoc、angptl4、cited2、isg20、ppp1r15a、prdx5和/或tgfbi。标志物例如akap12(a-kinase anchoring protein 12,gene id:9590)、aldoc(aldolase,fructose-bisphosphate c,gene id:230)、angptl4(angiopoietin like 4,gene id:51129)、cited2(cbp/p300 interacting transactivator with glu/asp rich carboxy-terminal domain 2,gene id:10370)、isg20(interferon stimulated exonuclease gene 20,gene id:3669)、ppp1r15a(protein phosphatase 1regulatory subunit 15a,gene id:23645)、prdx5(peroxiredoxin 5,gene id:25824)、tgfbi(transforming growth factor beta induced,gene id:7045);包括基因及其编码的蛋白及其同源物,突变,和同等型。该术语涵盖全长,未加工的标志物,以及源自细胞中加工的任何形式的标志物。该术语涵盖标志物的天然发生变体(例如剪接变体或等位变体)。gene id可在https://www.ncbi.nlm.nih.gov/gene/获得。
54.术语“受试者”意指任何动物,还指人类和非人类的动物。术语“非人类的动物”包括所有脊椎动物,例如,哺乳动物,如非人灵长类动物(特别是高等灵长类动物)、绵羊、狗、
啮齿类动物(如小鼠或大鼠)、豚鼠、山羊、猪、猫、兔、牛、和任何家畜或宠物;以及非哺乳动物,如鸡,两栖类,爬行动物等。
55.标志物可以在任何水平上差异地存在,但是一般以如下的水平存在,所述水平增加了至少5%、至少10%、至少15%、至少20%、至少25%、至少30%、至少35%、至少40%、至少45%、至少50%、至少55%、至少60%、至少65%、至少70%、至少75%、至少80%、至少85%、至少90%、至少95%、至少100%、至少110%、至少120%、至少130%、至少140%、至少150%、或更多;或一般以如下的水平存在,所述水平减少了至少5%、至少10%、至少15%、至少20%、至少25%、至少30%、至少35%、至少40%、至少45%、至少50%、至少55%、至少60%、至少65%、至少70%、至少75%、至少80%、至少85%、至少90%、至少95%、或100%(即不存在)。
56.优选地,标志物以具有统计显著性(即p值小于0.05和/或q值小于0.10,如使用韦尔奇氏t检验(welch's t-test)或wilcoxon秩和检验(wilcoxon's rank-sum test)所确定的水平差异地存在。
57.在本发明中,术语“样本”或“测试样本”指获自或衍生自目的个体的生物试样,生物试样的供给源可以是新鲜、冷冻和/或保存的器官或组织样品或从活检或引物产生的固体组织;血液或任意血液组分。术语“样本”或“测试样本”包括在其获得后以任意方式操作过的生物样本,如通过试剂处理、稳定化、或针对某些成分(如蛋白质或多核苷酸)富集、或包埋在用于切片目的的半固体或固体基质中。在本发明的一实施例中,将组织组分用作试样。
58.引物、探针、抗体、肽、适配体
59.本文使用的术语“引物”是识别靶基因序列的短核酸序列的链,其包括一对正反向引物。具体地,所述“引物”包括一对提供特异性和灵敏度的分析结果的引物。引物被认为在用于扩增靶基因序列时提供高度特异性,但它不引起与靶基因序列不一致或互补的非靶序列的扩增。
60.本文使用的术语“探针”是指特异性结合至样品中待检测的靶标的物质。通过所述结合,探针可确定样品中靶标的存在。只要它通常用于本领域中,任何探针都可用于本公开内容。特别地,所述探针可以是pna(肽核酸)、lna(锁核酸)、肽、多肽、蛋白、rna或dna,最优选pna。具体地,所述探针是一种生物材料,其可以来自有机体或可以体外合成、或是其模拟物。例如,所述探针可以是酶、蛋白、抗体、微生物、动物或植物细胞或器官、神经元、dna或rna。dna可包括cdna、基因组dna和寡核苷酸。同样地,基因组rna、mrna和寡核苷酸可落入rna的范围内。蛋白的实例包括抗体、抗原、酶和肽。
61.本文使用的术语“反义”是指具有核苷酸碱基序列和亚基-亚基骨架的寡聚体,所述骨架允许反义寡聚体通过watson-crick碱基配对与rna中的靶序列杂交以在靶序列中形成rna:寡聚体异源双链核酸分子。
62.本文所使用的术语“抗体”是本领域众所周知的,是指针对抗原位点的特异性免疫球蛋白。本发明中的抗体是指与本发明的标志物蛋白特异性结合的抗体,可以根据本领域中的常规方法来制造抗体。抗体的形式包括多克隆抗体或单克隆抗体、抗体片段(诸如fab、fab'、f(ab')2和fv片段)、单链fv(scfv)抗体、多特异性抗体(诸如双特异性抗体)、单特异性抗体、单价抗体、嵌合抗体、人源化抗体、人抗体、包含抗体的抗原结合位点的融合蛋白,
以及包含抗原结合位点的任何其他修饰的免疫球蛋白分子,只要该抗体表现出所需的生物结合活性。
63.本文所使用的术语“肽”具有与靶物质高度结合的能力,并且在热处理/化学处理期间不会发生变性。而且,由于其尺寸小,可以通过将其附接到其它蛋白上而用作融合蛋白。具体而言,因为可以特异性地附接到高分子蛋白链上,它可以用作诊断试剂盒和药物递送物质。
64.本文所使用的术语“适配体”是指一种由特定类型的单链核酸(dna、rna或修饰的核酸)组成的多核苷酸,所述单链核酸自身具有稳定的三级结构,并且具有能够以高亲和力和特异性与靶分子结合的特性。如上所述,由于适配体可以像抗体那样特异性结合抗原性物质,但比蛋白更稳定并具有简单的结构,并且是由易于合成的多核苷酸组成,因此可以代替抗体来使用
65.试剂盒
66.本公开内容提供了用于诊断卵巢癌或评估卵巢癌预后风险的试剂盒,其包含用于诊断卵巢癌或评估卵巢癌预后风险帕金森病的组合物。例如,所述试剂盒可以是rt-pcr试剂盒、dna芯片试剂盒、elisa试剂盒、蛋白芯片试剂盒、快速试剂盒、或mrm(多反应监测)试剂盒。
67.例如,所述诊断试剂盒可进一步包含反转录聚合酶链式反应所必需的元件。rt-pcr试剂盒包含一对特异性针对编码标记物蛋白的基因的引物。各引物是具有特异性针对所述基因的核酸序列的序列的核苷酸,其长度可为约7至50bp,更特别为约10至39bp。此外,所述试剂盒可进一步包含特异性针对对照基因的核酸序列的引物。此外,所述rt-pcr试剂盒可包含测试管或合适的器皿、反应缓冲液(不同ph值和镁浓度)、脱氧核苷酸(dntp)、酶(例如taq聚合酶和反转录酶)、脱氧核糖核酸酶抑制剂、核糖核酸酶抑制剂、depc-水、和无菌水。
68.此外,本公开内容的诊断试剂盒可包含用于操作dna芯片所必需的元件。所述dna芯片试剂盒可包含与基因或cdna或相当于其片段的寡核苷酸结合的底物、及用于构建荧光标记的探针的试剂、药剂和酶。此外,所述底物可包含对照基因或cdna或相当于其片段的寡核苷酸。
69.在一些实施方案中,本公开内容的试剂盒可包含用于进行elisa所必需的元件。所述elisa试剂盒可包含特异性针对蛋白的抗体。所述抗体具有针对标记物蛋白的高选择性和亲合力,与其他蛋白无交叉反应性,并且可以是单克隆抗体、多克隆抗体或重组抗体。此外,所述elisa试剂盒可包含特异性针对对照蛋白的抗体。此外,所述elisa试剂盒可进一步包含能够检测被结合的抗体的试剂,例如,标记的第二抗体、发色团、酶(例如,与抗体缀合)、及其底物或能够结合所述抗体的物质
70.系统、计算机可读存储介质
71.应当理解,本文使用的“系统”、“装置”、“单元”是用于区分不同级别的不同组件、元件、部件、部分或装配的一种方法。然而,如果其他词语可实现相同的目的,则可通过其他表达来替换所述词语。
72.所属技术领域的技术人员知道,本发明可以实现为设备、方法或计算机程序产品。因此,本公开可以具体实现为以下形式,即:可以是完全的硬件、也可以是完全的软件(包括
固件、驻留软件、微代码等),还可以是硬件和软件结合的形式,本文一般称为“单元”或“系统”。此外,在一些实施例中,本发明还可以实现为在一个或多个计算机可读介质中的计算机程序产品的形式,该计算机可读介质中包含计算机可读的程序代码。
73.可以采用一个或多个计算机可读的介质的任意组合。计算机可读介质可以是计算机可读信号介质或者计算机可读存储介质。计算机可读存储介质例如可以是但不限于电、磁、光、电磁、红外线、或半导体的系统、装置或器件,或者任意以上的组合。计算机可读存储介质的更具体的例子(非穷举的列表)包括:具有一个或多个导线的电连接、便携式计算机磁盘、硬盘、随机存取存储器(ram)、只读存储器(rom)、可擦式可编程只读存储器(eprom或闪存)、光纤、便携式紧凑磁盘只读存储器(cd-rom)、光存储器件、磁存储器件,或者上述的任意合适的组合。在本文件中,计算机可读存储介质可以是任何包含或存储程序的有形介质,该程序可以被指令执行系统、装置或者器件使用或者与其结合使用。
74.计算机可读的信号介质可以包括在基带中或者作为载波一部分传播的数据信号,其中承载了计算机可读的程序代码。这种传播的数据信号可以采用多种形式,包括但不限于电磁信号、光信号或上述的任意合适的组合。计算机可读的信号介质还可以是计算机可读存储介质以外的任何计算机可读介质,该计算机可读介质可以发送、传播或者传输用于由指令执行系统、装置或者器件使用或者与其结合使用的程序。
75.计算机可读介质上包含的程序代码可以用任何适当的介质传输,包括但不限于无线、电线、光缆、rf等等,或者上述的任意合适的组合。
76.可以以一种或多种程序设计语言或其组合来编写用于执行本发明操作的计算机程序代码,所述程序设计语言包括面向对象的程序设计语言—诸如java、smalltalk、c++,还包括常规的过程式程序设计语言诸如“c”语言或类似的程序设计语言。程序代码可以完全地在用户计算机上执行、部分地在用户计算机上执行、作为一个独立的软件包执行、部分在用户计算机上部分在远程计算机上执行、或者完全在远程计算机或服务器上执行。在涉及远程计算机的情形中,远程计算机可以通过任意种类的网络包括局域网(lan)或广域网(wan)连接到用户计算机,或者,可以连接到外部计算机(例如利用因特网服务提供商来通过因特网连接)。
77.以下所举实例是为了对本发明的的某些优选实施方案和本发明的某些方面进行描述,并不应解释为限定本发明的范围。以下实例结合附表和附图对本发明所述实施方案做进一步的详细说明。
78.实施例1构建卵巢癌预后模型
79.一、实验方法
80.1、数据集来源和预处理
81.在基因表达综合数据库(geo)和癌症基因组图谱数据库(tcga)中搜索公共基因表达数据和完整的临床注释。对于tcga中的数据集,基因表达的rna测序数据(fpkm值)和临床信息从ucsc xena(https://gdc.xenahubs.net)下载。然后将fpkm值转化为每千碱基百万(tpm)值的转录本。从geo数据库(http://www.ncbi.nlm.nih.gov/geo/)下载gse17260及gse32062的基因表达数据,并利用注释文件对其注释,多个探针对应同一个基因的取平均值作为其表达量,然后获得基因表达矩阵文件。将两个基因表达矩阵文件合并到一个文件中,并使用“sva”r包对来自两个不同数据集的表达数据进行批量归一化。最后,获得了一个
标准化的基因表达矩阵文件。其中,tcga数据集作为发现队列,geo数据集作为验证队列,数据集的详细信息见表1。缺氧相关基因列表从分子特征数据库的标志基因组(hallmark gene sets)中获得,共有191个基因被纳入分析,它们都对低氧水平有反应。
82.表1本研究中数据集的基本信息
[0083][0084][0085]
2、缺氧基因signature的构建与验证
[0086]
在发现队列中,单因素cox回归分析用于确定与预后相关的缺氧基因(p《0.01)。利用r中的“glmnet”软件包,通过cox回归和lasso方法构建一个预测ov患者预后的缺氧基因signature。在这个分析中,采用了套索惩罚,同时考虑收缩和变量选择。通过10次交叉验证,确定了lambda惩罚参数的最佳值。本发明使用以下公式计算ov患者的风险评分:
[0087][0088]
根据中位评分将患者分为高危组和低危组。另外,在验证队列中采用相同的公式计算风险评分。
[0089]
3、生存分析
[0090]
使用r中的“survival”和“survminer”软件包,通过kaplan-meier分析比较高、低缺氧风险组的os。采用单因素cox分析确定潜在的预后因素,多因素cox分析确定风险评分作为ov患者os的独立危险因素。通过“timeroc”r软件包生成roc曲线,以验证风险模型预测患者生存期的准确性。
[0091]
4、统计分析
[0092]
所有统计均使用r软件(版本3.6.3;https://www.r-project.org)。wilcox检验用于筛选统计差异表达的基因。绘制kaplan-meier曲线,用log-rank检验组间os的显著性差
异。还进行了单因素和多因素cox比例风险回归分析,以了解风险评分与os之间的关系。采用roc分析评估风险评分在预测预后的敏感性和特异性,roc曲线下面积(auc)是判断预后准确性的指标。p值小于0.05为所有分析具有统计学意义。
[0093]
二、实验结果
[0094]
1、本发明所涉及基因signature预后模型的构建
[0095]
对191个缺氧基因进行单因素cox回归分析,筛选出与ov预后显着相关的九个基因如图1所示。之后,通过对九个特征基因进行lasso cox回归分析。构建了由akap12、aldoc、angptl4、cited2、isg20、ppp1r15a、prdx5和tgfbi组成的八基因风险模型,如图2所示。使用lasso算法获得的系数计算tcga训练队列的风险得分,公式如下:
[0096]
风险得分=(0.045
×
akap12表达水平)+(0.099
×
aldoc表达水平)+(0.109
×
angptl4表达水平)+(0.096
×
cited2表达水平)-(0.306
×
isg20表达水平)+(0.046
×
ppp1r15a表达水平)-(0.169
×
prdx5表达水平)+(0.045
×
tgfbi表达水平)
[0097]
2、风险信号的预后价值
[0098]
热图结果表明,八个缺氧基因中有六个是在高风险评分组中高表达,这意味着高风险患者倾向于发展缺氧的微环境。本发明的数据还显示,高风险组的死亡率显着高于低风险组,如图3、4所示。此外进行kaplan-meier生存分析以评估ov缺氧信号的预后价值,结果高风险评分组患者的生存时间明显短于低风险评分组,如图5所示。高缺氧风险评分与发现队列中的os差有关,这已由验证队列进一步验证。
[0099]
3、缺氧signature显示出强大的预后评估能力
[0100]
为了评估低氧signature在1年,3年和5年生存率中的预测效率,本发明利用发现队列和验证队列数据集的数据执行了接收的操作特征(roc)曲线。roc曲线下的面积(auc)在1年时分别为0.67、3年时为0.64和5年时为0.71,表明预测值较高。geo数据集对此进行了进一步验证,如图6所示。然后将单因素和多因素cox分析缺氧风险信号用于评估ov患者os的的独立预后价值。单因素分析表明,风险评分高与os差有关。多因素分析显示,风险评分与ov患者的os明显较差相关,可作为ov的独立预后因素。这些已通过geo数据库进行了验证,如图7、8所示。
[0101]
实施例2 real time pcr检测卵巢癌组织样本中目的基因表达量的变化
[0102]
一、实验目的
[0103]
用sybr green i嵌合荧光法检测卵巢癌组织样本中目的基因mrna转录水平的变化情况。
[0104]
二、实验材料
[0105]
1、样本清单
[0106]
实验室提供10份卵巢组织样本,其中5份卵巢癌组织样本,5份癌旁对照组织:
[0107]
表2样本清单
[0108]
样本序号样本名称样本序号样本名称1c16e12c27e23c38e34c49e4
5c510e5
[0109]
2、实验主要试剂
[0110]
表3使用试剂清单
[0111][0112]
3、实验主要仪器
[0113]
表4使用仪器清单
[0114]
仪器名称仪器型号厂家电动组织匀浆器dy89
‑‑
11宁波新芝生物科技股份有限公司离心机centrifuge 5424reppendorfnanovue plus28956057biochrom ltd荧光定量pcr仪abi7300applied biosystems
[0115]
三、实验方法
[0116]
1.引物设计
[0117]
1.1 real time pcr检测目的基因引物。以下引物在博迈德公司合成。
[0118]
表5引物序列
[0119][0120][0121]
2.实验过程
[0122]
2.1提取样本总rna
[0123]
①
将1ml trizol在超净台里加入至玻璃匀浆瓶中(提前将匀浆瓶用烘箱180度烘4个小时),将匀浆瓶按到仪器上,称取50—100mg的组织放入玻璃匀浆瓶内,将转速调至1500转左右,开始在冰水浴中进行匀浆,每研磨30s,停止30s,反复3-4次即可。样品体积不应超过trizol体积10
℅
。
[0124]
②
将加入trizol的样品在室温(15-30℃)放置10min,使核酸蛋白复合物完全分
离。
[0125]
③
1ml trizol加入200μl氯仿,剧烈振荡2min,每隔1分钟再晃动两下,5—6次后,再静止7min。
[0126]
④
4℃,12000rpm,离心15min。样品分为三层:底层为黄色有机相,上层为无色水相和一个中间层。rna主要在水相中,水相体积约为所用trizol的60
℅
。
[0127]
⑤
把上层水相转移到新的ep管中(约400ul,尽量不要吸取到中间层以免污染)。加入500μl异丙醇,室温放置10min。
[0128]
⑥
制备75%乙醇,置于冰盒中预冷。
[0129]
⑦
4℃,12000rpm,离心15min,离心后在管底出现白色沉淀。用移液器小心移去上清。
[0130]
⑧
加入1ml 75
℅
冷乙醇,震荡洗涤沉淀。4℃,7500rpm,离心5min,小心弃掉上清。
[0131]
⑨
将ep管倒扣于滤纸上吸去多余的水分,并用10μl枪头小心吸取管内的液体(枪头不要接触rna),将ep管室温放置5min(时间太久,过于干燥会使rna活性降低),rna变透明;
[0132]
⑩
加入40μl无rnase的水(depc水),用nanodrop检测od值与浓度,在管上做好标记;
[0133]
置于-80℃冰箱保存。
[0134]
2.2逆转录合成mrnacdna
[0135]
采用fastking cdna第一链合成试剂盒(货号:kr116)进行mrna反转录,首先去除基因组dna反应,在试管中加入5
×
gdna buffer 2.0ul,totalrna 1ug,加rnase free ddh2o使总体积至10ul,水浴锅中42℃加热3min,再将10
×
king rt buffer 2.0ul,fastking rt enzyme mix 1.0ul,fq-rt primer mix 2.0ul,rnase free ddh2o 5.0ul,混合后加入上述试管中一起混合共20ul,水浴锅中42℃加热15min,95℃加热3min,合成的cdna需要长期保存时,请于-20℃或更低温度保存。
[0136]
2.3 realtimepcr
[0137]
2.3.1 mrna荧光定量检测
[0138]
2.3.1.1仪器及分析方法
[0139]
用abi 7300型荧光定量pcr仪,采用2-△△
ct
法进行数据的相对定量分析。
[0140]
2.3.1.2操作过程如下:
[0141]
(一)反应体系:用superreal premix plus(sybr green)(货号:fp205),进行扩增,实验操作按产品说明书进行。realtime反应体系为:
[0142]
表6 realtime反应体系
[0143]
[0144][0145]
(二)扩增程序为:95
°
15min,(95℃10sec,55℃30sec,72℃32sec)
×
40个循环,95℃15sec,60℃60sec,95℃15sec)。
[0146]
(三)引物筛选
[0147]
将各样本cdna混合后,以此为模板进行10倍稀释,稀释后样品各取2μl作模板,分别用目的基因引物和内参基因引物进行扩增,同时在60-95℃进行溶解曲线分析,根据扩增效率高和溶解曲线单峰原则进行引物筛选。
[0148]
(四)样品realtimepcr检测
[0149]
将各样品cdna 10-20倍稀释后取2μl作模板,分别用目的基因引物和内参基因引物进行扩增(见表7)。同时在60-95℃进行溶解曲线分析。
[0150]
表7样品realtimepcr检测设计
[0151]
模板样品cdna样品cdna重复检测孔道数33引物目的基因引物内参基因引物
[0152]
四、实验结果
[0153]
1.rna浓度检测结果及1.5%琼脂糖rna电泳检测结果
[0154]
表8 rna浓度及纯度结果
[0155]
[0156][0157]
注:rna溶于水中会导致a260/280比值偏低
[0158]
注:a:浓度未达标准;b:a260/a280不合格;c:电泳图不合格;h:合格
[0159]
样本评判标准:
[0160]
(1)浓度》30ng/ul
[0161]
(2)1.8《a260/a280《2.0
[0162]
(3)电泳图可以看到较清晰三条带(第三条可能会看不到)
[0163]
(4)rt-pcr后产物电泳图可以看到较清晰的一条带
[0164]
表9电泳上样情况
[0165]
[0166][0167]
pcr产物电泳图如图9所示,图中pcr产物电泳图的顺序为:marker、c1、e1、c2、e2、c3、e3、c4、e4、c5、e5共10个样本。根据跑胶结果最终选取c1、e1、c2、e2、c3、e3、c4、e4、c5、e510个样本进行实时定量实验。mark:dna marker:dm2000,从下往上依次100,250,500,750,1000和2000bp,其中750bp为亮带。
[0168]
2.各样品realtimepcr检测结果及分析
[0169]
2.1各样本实时扩增曲线图和样本扩增产物溶解曲线图
[0170]
内参gapdh基因实时扩增曲线图及产物溶解曲线图如图10所示;内参actb基因实时扩增曲线图及产物溶解曲线图如图11所示;akap12基因实时扩增曲线图及产物溶解曲线图如图12所示;aldoc基因实时扩增曲线图及产物溶解曲线图如图13所示;angptl4基因实时扩增曲线图及产物溶解曲线图如图14所示;cited2基因实时扩增曲线图及产物溶解曲线图如图15所示;ppp1r15a基因实时扩增曲线图及产物溶解曲线图如图16所示;prdx5基因实时扩增曲线图及产物溶解曲线图如图17所示。
[0171]
2.2各样品相对定量分析结果
[0172]
根据realtimepcr原始检测结果,按照相对定量计算公式,即
[0173][0174]
计算出各样品的目的基因相对定量结果,即其他各个样品相对于对照样品,目的基因mrna转录水平的差异。
[0175]
如图18所示,与对照组相比,aldoc、cited2、prdx5在卵巢癌组织中显著上调,akap12、angptl4、ppp1r15a在卵巢癌组织中显著下调。
[0176]
以上所述实施例仅表达了本发明的几种实施方式,其描述较为具体和详细,但并不能因此而理解为对发明专利范围的限制。应当指出的是,对于本领域的普通技术人员来说,在不脱离本发明构思的前提下,还可以做出若干变形和改进,这些都属于本发明的保护
范围。因此,本发明专利的保护范围应以所附权利要求为准。
