no.1084所示的序列组成。
9.上述探针组中,各条探针独立包装或混合包装。
10.在本发明具体的实施方式中,所述探针组中的各条探针是针对上述遗传性胆红素代谢异常和/或肝内胆汁淤积症的10个易感基因的特定序列进行设计,各条探针单独合成并独立包装,在进行检测时混合后加入。
11.包含上述探针组的试剂盒也在本发明的保护范围之内。
12.在本发明试剂盒中还包括用于检测或辅助检测遗传性胆红素代谢异常和/或肝内胆汁淤积症相关基因变异的其他试剂,例如连接酶、聚合酶、磁珠、缓冲液等等。
13.本发明进一步提供了上述探针组或试剂盒在如下任一所述的应用:
14.1)在检测或辅助检测遗传性胆红素代谢异常和/或肝内胆汁淤积症相关基因变异中的应用;
15.2)在制备检测或在辅助检测遗传性胆红素代谢异常和/或肝内胆汁淤积症相关基因变异的产品中的应用;
16.3)在制备检测或辅助检测遗传性胆红素代谢异常和/或肝内胆汁淤积症的产品中的应用。
17.本发明进一步提供了一种检测或辅助检测遗传性胆红素代谢异常和/或肝内胆汁淤积症相关基因变异的方法,包括如下步骤:
18.提取待测样本的基因组dna,构建dna文库;
19.将上述探针组或试剂盒中各条探针混合后与所述dna文库靶向捕获,获得靶向捕获文库;
20.对所述靶向捕获文库进行二代测序获得测序数据,对测序数据进行分析得到待测样本的遗传性胆红素代谢异常和/或肝内胆汁淤积症相关基因的变异结果。
21.上述方法中,所述基因组dna为外周血细胞基因组dna。
22.在本发明中检测或辅助检测遗传性胆红素代谢异常和/或肝内胆汁淤积症相关基因变异的方法中,dna文库的构建、靶向捕获文库的获得和二代测序均可以采用本领域常规方法进行。
23.本发明具体的实施方式中,所述dna文库的构建包括dna打断、末端修复和3’端加“a”、接头连接和纯化、pre-pcr反应及纯化。
24.本发明具体的实施方式中,所述靶向捕获文库的获得包括dna文库浓缩、探针杂交、捕获目标区域dna文库、poster-pcr反应等。
25.在本发明具体的实施方式中,所述对测序数据进行分析得到待测样本的遗传性胆红素代谢异常和/或肝内胆汁淤积症相关基因的变异结果是通过序列筛选和比对等方法并进行生物学信息分析,得到了待测样本的遗传性胆红素代谢异常和/或肝内胆汁淤积症相关基因的全部外显子及内含子剪切区的单核苷酸多态性(snp)和/或拷贝数变异(cnv)。
26.上述探针组、试剂盒和方法中,所述遗传性胆红素代谢异常和/或肝内胆汁淤积症相关基因变异为遗传性胆红素代谢异常和/或肝内胆汁淤积症相关基因的全部外显子及内含子剪切区的单核苷酸多态性(snp)和/或拷贝数变异(cnv)。本发明遗传性胆红素代谢异常和/或肝内胆汁淤积症相关基因变异的参考基因组为人基因组hg19,即是相对于人基因组hg19对应基因发生的变异,所述人基因组hg19为人类(homo sapiens)标准参考基因组
(grch37/hg19)。
27.上述探针组、试剂盒和方法中,所述遗传性胆红素代谢异常相关基因为如下易感基因中至少一种:ugt1a1基因、abcc2基因、slco1b1基因和slco1b3基因;所述肝内胆汁淤积症相关基因为如下易感基因中至少一种:atp8b1基因、abcb11基因、abcb4基因、jag1基因、slc25a13基因和serpina1基因。
28.上述探针组、试剂盒和方法中,所述遗传性胆红素代谢异常为如下疾病中至少一种:gilbert综合征(对应ugt1a1基因)、crigler-najjar综合征(对应ugt1a1基因)、dubin-johnson综合征(对应abcc2基因)和rotor综合征(对应slco1b1和slco1b3基因);所述肝内胆汁淤积症为如下疾病中至少一种:良性复发型肝内胆汁淤积症(对应atp8b1基因)、进行性家族性肝内胆汁淤积症1型(对应atp8b1基因)、进行性家族性肝内胆汁淤积症2型(对应abcb11基因)、进行性家族性肝内胆汁淤积症3型(对应abcb4基因)、alagille综合征(对应jag1基因)、citrin缺乏症(对应slc25a13基因)和α1-抗胰蛋白酶缺乏症(对应serpina1基因)。
29.本发明的有益效果如下:
30.本发明检测遗传性胆红素代谢异常和/或肝内胆汁淤积症相关基因变异的方法集成了对遗传性胆红素代谢异常和/或肝内胆汁淤积症具有检测意义的10个易感基因的全部外显子及内含子剪切区的snp/cnv,具有如下优点:(1)检测通量高,一次反应可检测10个易感基因的全部外显子及内含子剪切区的snp/cnv;(2)特异性和灵敏度高;测序覆盖度100%,深度》30
×
;(3)基因检测结果判读明确客观;(4)成本较低,操作简便,易于普及。本发明方法适用于:(1)临床不明原因反复胆红素代谢异常和肝内胆汁淤积患者的鉴别诊断;(2)对胆红素代谢异常及肝内胆汁淤积易感人群,尤其是对具有遗传性肝病家族遗传史的人群具有一定的辅助诊断和提示作用;(3)可以简便高效的对普通人群进行遗传性胆红素代谢异常及肝内胆汁淤积症的群体性筛查。
附图说明
31.下面结合附图对本发明的具体实施方式作进一步详细的说明。
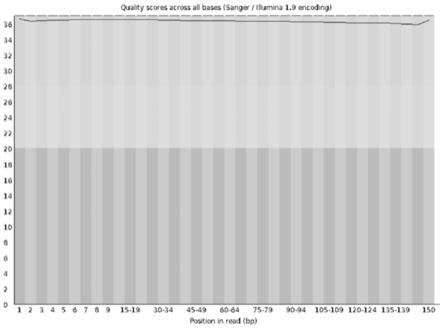
32.图1是样本g365测序碱基质量分布图。测序数据的质量均分布在q30(原始数据中质量值》=30的碱基)以上。
33.图2是样本g365碱基测序深度积累图。其中,目标区域不同测序深度(均一化处理,平均深度为1)的累积占比,比如深度为0.2时纵坐标为99.97,代表99.97%的目标区域测序深度大于平均深度的20%。
具体实施方式
34.为了更清楚地说明本发明,下面结合优选实施例和附图对本发明做进一步的说明。附图中相似的部件以相同的附图标记进行表示。本领域技术人员应当理解,下面所具体描述的内容是说明性的而非限制性的,不应以此限制本发明保护范围。
35.下述实施例中所使用的材料、试剂等,如无特殊说明,均可从商业途径获得。所有引物纯度达到电泳级(page)或hplc级,不含杂带。下述实施例中所使用的实验方法如无特殊说明,均为二代测序常规方法。
36.下述实施例中涉及的临床血液样本来自首都医科大学附属北京友谊医院。血液样本采集后,立即放入-80℃冰箱中保存。
37.实施例1用于检测遗传性胆红素代谢异常和/或肝内胆汁淤积症相关基因变异的试剂盒及使用方法
38.一、用于检测遗传性胆红素代谢异常和/或肝内胆汁淤积症相关基因变异的试剂盒
39.本发明用于检测遗传性胆红素代谢异常和/或肝内胆汁淤积症相关基因变异的试剂盒包括与良性复发型肝内胆汁淤积症、进行性家族性肝内胆汁淤积症1型、进行性家族性肝内胆汁淤积症2型、进行性家族性肝内胆汁淤积症3型、gilbert综合征、crigler-najjar综合征、alagille综合征、dubin-johnson综合征、rotor综合征、citrin缺乏症和α1-抗胰蛋白酶缺乏症等遗传性胆红素代谢异常和肝内胆汁淤积症相关的10个易感基因(见表1)相匹配的1084条探针,序列如sed id no.1-sed id no.1084所示(所述探针所对应的捕获区域如表2所示)。
40.其中,遗传性胆红素代谢异常和胆汁淤积症易感基因对应的遗传代谢性肝病如表1所示:
41.表1遗传性胆红素代谢异常和胆汁淤积症易感基因对应的疾病信息
[0042][0043][0044]
表2探针对应的捕获区域信息
[0045]
[0046]
[0047]
[0048]
[0049]
[0050]
[0051][0052]
注:表中探针编号对应探针的序列编号,chr18:55315698-55315797表示染色体编号chr18,区域位置55315698-55315797,其他同。对应的为人类(homo sapiens)标准参考基因组(grch37/hg19)。
[0053]
二、用于检测遗传性胆红素代谢异常和/或肝内胆汁淤积症相关基因变异的试剂盒的使用方法(即检测遗传性胆红素代谢异常和/或肝内胆汁淤积症相关基因变异的方法)
[0054]
1.基因组dna提取
[0055]
取待测样本的全血混匀后使用qiaamp dna blood mini kit(qiagen公司,usa)提取获得外周血细胞基因组dna。dna浓度使用qubit dsdna hs assay kit与qubit fluorometer(life technologies公司,usa)机器定量。dna纯度(a260/280与a260/230)使用nanodrop-2000(thermo fisher scientific公司,usa)鉴定。
[0056]
2.dna文库构建
[0057]
基因组dna使用bioruptor pico打断仪打断成150bp-200bp的dna片段,测序文库经one kit-box1(for ilm)(艾吉泰康生物科技(北京)有限公司)制备。
[0058]
2.1末端修复、3’端加“a”,体系如表3所示。
[0059]
表3末端修复、3’端加“a”的体系
[0060]
组分用量150bp-200bp的dna片段35μl10
×
era buffer(10
×
era缓冲液)5μl5
×
era-tailing enzyme mix(末端修复酶混合液)10μl总体积50μl
[0061]
体系使用移液器吹打混匀并避免剧烈震荡混匀,将pcr管放回冰盒上备用;运行pcr程序,设置pcr仪参数如下:热盖85℃,4℃1分钟,20℃30分钟,65℃30分钟,4℃保温,得到末端修复、3’端加“a”后的产物。
[0062]
2.2接头连接,体系如表4所示。
[0063]
表4接头连接的体系
[0064]
组分用量步骤2.1得到的末端修复、3’端加“a”后的产物50μladapter for illumina(illumina通用接头,15μm)5μlnuclease-free water(无核酸酶水)15μl5
×
ligation buffer(连接缓冲液)20μlligase(连接酶)10μl总体积100μl
[0065]
体系轻轻吸打混匀6次,避免产生气泡,然后短暂离心;运行pcr程序(要求无热盖),设置pcr仪参数如下:20℃15分钟,4℃保温得到接头连接后的产物并立即进行下一步实验。
[0066]
2.3连接后纯化,体系如表5所示。
[0067]
纯化使用磁珠为agencourt ampure xp,将agencourt ampure xp磁珠拿至室温,震荡混匀备
[0068]
表5纯化体系
[0069]
组分用量步骤2.2得到的接头连接后的产物100μlagencourt ampure xp磁珠80μl总体积180μl
[0070]
体系轻轻吸打混匀6次,室温静置孵育5-15分钟,将pcr管置于磁力架上3分钟使溶液澄清;移除上清,pcr管继续放置在磁力架上,向pcr管内加入200μl 80%乙醇溶液,静置30秒;移除上清,再向pcr管内加入200μl 80%乙醇溶液,静置30秒后彻底移除上清;室温静置3-5分钟,使残留乙醇彻底挥发;加入22μl的无核酸酶水,把pcr管从磁力架取下,轻轻吸打重悬磁珠,避免产生气泡,室温静置2分钟;将pcr管置于磁力架上2分钟使溶液澄清;用移液器吸取20μl上清液,转移到新的pcr管中(置于冰盒上)得到纯化后的产物,在反应管上标记样本编号,准备下一步反应。
[0071]
2.4 pre-pcr反应,体系如表6所示。
[0072]
预先从-20℃保存的试剂盒中取出pcr master mix、tpe1.0和tpe2.0试剂,置于冰盒上溶解,混匀后置于冰上待用并操作。
[0073]
表6 pre-pcr反应体系
[0074]
组分用量步骤2.3得到的纯化后的产物20μlpcr master mix(pcr反应混合物)25μltpe1.0(p1端index引物,20μm)2.5μltpe2.0index-8(p2端index引物,20μm)2.5μl总体积50μl
[0075]
体系使用移液器轻轻吹打混匀,然后短暂离心;运行pcr程序,设置pcr仪参数如下:热盖105℃,98℃2分钟;98℃20秒,60℃30秒,72℃30秒共7个循环;72℃1分钟,4℃保温得到pre-pcr反应后的产物并立即进行纯化。
[0076]
2.5 pre-pcr反应后纯化
[0077]
向步骤2.4得到的pre-pcr反应后的pcr管中加入50μl agencourt ampure xp磁珠,用移液器吹打混匀,避免产生气泡;室温孵育5-15分钟,把pcr管置于磁力架上3分钟使溶液澄清;移除上清,pcr管继续放置在磁力架上,向pcr管内加入200μl 80%乙醇溶液,静置30秒;移除上清,再向pcr管内加入200μl 80%乙醇溶液,静置30秒后彻底移除上清;室温静置5分钟,使残留乙醇彻底挥发;加入30μl的无核酸酶水,将离心管从磁力架取下,使用移液器,轻轻吸打重悬磁珠;室温静置2分钟,将200μl pcr管置于磁力架上2分钟使溶液澄清;用移液器将上清液转移到新的200μl pcr管中(置于冰盒上)得到dna文库,在反应管上标记好样本号,准备下一步反应;取1μl样品使用3.0fluorometer(qubit dsdna hs assay kit)进行dna文库浓度测定,记录dna文库浓度,文库浓度》25ng/μl;取1μl样品使用agilent 2100 bioanalyzer system(agilent dna 1000kit)进行片段长度测定,dna文库长度在270bp-320bp间。
[0078]
3.靶向捕获文库构建
[0079]
3.1dna文库浓缩,体系如表7所示。
[0080]
预先从-20℃保存的捕获试剂盒中,取出表5中所示block试剂,放置于冰盒上溶解,溶解混匀后置于冰上或4℃待用。
[0081]
表7浓缩体系
[0082]
组分用量
步骤2.5得到的dna文库750nghyb human block5μlada-block for ilm(pe1.0&2.0-8bp)2μl
[0083]
体系混匀后标记为b管,将b管放入真空浓缩离心机,打开pcr管盖,启动离心机,打开真空泵开关,开始浓缩。将b管浓缩至体积小于10μl,之后用无核酸酶水补足至10μl,轻轻吸打混匀,短暂离心后置于冰上待用。
[0084]
3.2探针制备与杂交
[0085]
探针制备包含模板合成和rna制备。模板合成采用的是高通量电化学合成方式。在一次性的芯片上,通过控制电信号的有无,来决定是否添加对应的碱基。合成的oligo混合在一起即pooling或混池的过程。质检合格的oligo pool产物用体外转录的方式合成rna探针。在合成时,使用生物素(biotin)标记的ntp和核苷酸类似物(如lna、pna)作为合成原料。核苷酸类似物可以提高探针和文库结合的tm值,使探针和文库之间的结合更稳固。带有生物素(biotin)标记的探针可以在后续实验时和链霉亲和素(streptavidin)包被的磁珠结合。探针设计深度为3x,设计好的探针序列会经过热力学稳定性评估,评估结果较差的区域,会补充设计探针,以达到理想的覆盖度、均一度和捕获效率。
[0086]
将one hyb buffer置于室温融化,混匀后置于50℃恒温混匀仪内预热,完全溶解后(无沉淀及浑浊物)每个反应取20μlone hyb buffer置于新的200μl pcr管内,盖好管盖,置于50℃恒温混匀仪孵育待用,标记为a管;取5μl rnase block与2μl rna探针置于200μl pcr管内,轻轻吸打混匀,短暂离心后置于冰上或4℃待用,标记为c管;从a管吸取13μl与b管10μl加入c管中震荡混匀,短暂离心后置于冰上待用。运行pcr程序,设置pcr仪参数如下:热盖85℃,80℃5分钟;98℃20秒,50℃保温孵育过夜,得到杂交后的产物。
[0087]
3.3捕获磁珠平衡
[0088]
捕获磁珠(cap beads)从4℃取出,涡旋震荡重悬,置于室温平衡30分钟;取50μl磁珠置于新的pcr管内,置于磁力架上1分钟使溶液澄清,移除上清;从磁力架上取下pcr管,加入200μl结合缓冲液(binding buffer)轻轻吸打数次混匀,重悬捕获磁珠;置磁力架上1分钟,移除上清;重复上述步骤两次,共清洗磁珠3次;从磁力架上取下pcr管,加入180μl结合缓冲液轻轻吸打6次重悬捕获磁珠得到平衡后的捕获磁珠待用。
[0089]
3.4捕获目标区域dna文库
[0090]
保持步骤3.2得到的杂交后的产物在pcr仪上,取180μl步骤3.3得到的平衡后的捕获磁珠加入到杂交后的产物中,用移液器吸打6次混匀,置于旋转混匀仪上室温结合30分钟;将pcr管置于磁力架上2分钟使溶液澄清,移除上清液;pcr管内加入200μl的清洗液1(wash buffer 1),轻轻吸打6次混匀,置于旋转混匀仪上清洗15分钟,然后短暂离心,将pcr管放于磁力架上2分钟,使溶液澄清,移除上清;加入200μl的50℃预热的one wash buffer 2,轻轻吸打6次混匀,置于恒温振荡混匀仪上50℃孵育10分钟;短暂离心,将pcr管放于磁力架上2分钟,移除上清。使用onewash buffer 2清洗2次,共计3次。最后一次彻底移除one wash buffer 2;保持样品在磁力架上,向pcr管内加入200μl 80%乙醇,静置30s后彻底移除乙醇溶液,室温晾干;向pcr管加入30μl无核酸酶水,从磁力架上取下pcr管,轻轻吸打6次重悬捕获磁珠,得到捕获目标区域dna文库待用。
[0091]
3.5 poster-pcr反应,体系如表8所示。
[0092]
预先从-20℃保存的试剂盒中取出post pcr buffer、post pcr primer(25μm,for ilm)试剂,放置冰上溶解,溶解混匀后置于冰上待用;取出post pcr polymerase短暂离心。
[0093]
表8 poster-pcr反应体系
[0094]
组分用量步骤3.4得到的捕获目标区域dna文库30μlpost pcr buffer(post pcr缓冲液)18μlpost pcr primer(post pcr引物,25μm,for ilm)1μlpost pcr polymerase(post pcr聚合酶)1μl总体积50μl
[0095]
将移液器调至40μl,轻轻吸打体系混匀6次,立即运行pcr程序,设置pcr仪参数如下:热盖105℃;95℃1分钟;98℃20秒,60℃30秒,72℃30秒共循环12次;72℃5分钟;4℃保温。pcr结束后向样品加入55μl agencourt ampure xp磁珠,用移液器轻轻吸打6次混匀;室温孵育5分钟,把pcr管置于磁力架上3分钟使溶液澄清;移除上清,pcr管继续置于磁力架上,加入200μl 80%乙醇,静置30秒;移除上清,再向pcr管内加入200μl 80%乙醇,静置30秒后彻底移除上清;室温放置5分钟,使得残留乙醇彻底挥发;加入25μl无核酸酶水,将pcr管从磁力架拿下,轻轻吹打混匀重悬磁珠,室温放置2分钟;将pcr管置于磁力架上2分钟;用移液器吸23μl上清液转移到1.5ml离心管,标记样品信息,获得靶向捕获文库。
[0096]
4.二代测序
[0097]
4.1测序
[0098]
从靶向捕获文库中取1μl样品使用qubit dsdna hs assay kit进行定量,记录文库浓度,文库浓度在1-20ng/μl;取1μl样品使用agilent 2100bioanalyzer system(agilent dna 1000kit)进行片段长度测定,文库长度在270bp-320bp间。
[0099]
靶向捕获文库在illumina nextseq cn500平台,使用nextseq cn500mid output v2kit(illumina公司)进行测序,得到原始测序数据。
[0100]
4.2数据处理
[0101]
序列筛选:原始测序数据去除低质量reads定义为n碱基(n表示无法确定碱基信息)的比例大于10%并且碱基质量值(base quality)小于5的碱基数占整个read的50%以上的reads。trimmomatic用于fastq质控。
[0102]
比对:测序数据与参考基因组进行比对。参考基因组是人基因组hg19,即人类(homo sapiens)标准参考基因组(grch37/hg19)。reads使用bwa算法映射人基因组hg19。
[0103]
生物学信息分析:clean data用于变异检测分析,应用varscan软件得到最终的注释文件。
[0104]
最终得到待测样本的遗传性胆红素代谢异常和/或肝内胆汁淤积症相关基因的全部外显子及内含子剪切区的单核苷酸多态性(snp)和/或拷贝数变异(cnv)情况。
[0105]
5.某家系不明原因肝功能异常者检测结果
[0106]
利用步骤1-4所述的方法对患者18c052625及患者父亲18c052626和患者母亲18c052627进行检测,结果显示患者18c052625检测出abcb11基因有复合杂合突变:c.1904t》a(p.v635e),为杂合错义突变,正常对照人群中该变异频率极低,符合pm2;多种生物信息
学蛋白功能预测软件为有害,符合pp3,经家系验证分析,患者母亲存在该杂合变异。c.2815-8a》g(splicing),为杂合剪接突变,正常对照人群中未见该变异,符合pm2;体内体外功能实验已明确会导致基因功能受损,符合ps3,经家系验证分析,该变异来自患者父亲。故该复合杂合变异判定为致病性变异,会导致进行性家族性肝内胆汁淤积症2型。同时检测出该患者slco1b3基因c.508_509del(p.m170fs)为杂合移码突变,该变异为零效变异,可能导致基因功能丧失,符合pvs1;正常对照人群中该变异频率极低,符合pm2;经家系验证分析,该变异来自患者母亲。但患者及其母亲slco1b1基因无致病性突变,故不会引起rotor综合征。
[0107]
具体的患者18c052625的检测结果如表9所示:
[0108]
表9患者18c052625的检测结果
[0109]
突变位置核苷酸改变氨基酸改变纯合/杂合致病性突变类型abcb11外显子22c.2815-8a》g-杂合(疑似)致病剪切区变异abcb11外显子16c.1904t》ap.v635e杂合(疑似)致病错义突变abcb11外显子13c.t1331cp.v444a纯合良性错义突变ugt1a1外显子1c.g211ap.g71r杂合未知错义突变abcb4外显子16c.a1954gp.r652g杂合良性错义突变slc25a13外显子4c.a328+6g-杂合未知剪切区变异abcc2外显子10c.g1249ap.v417i杂合良性错义突变slco1b3外显子5c.t334gp.s112a杂合良性错义突变slco1b3外显子5c.c335ap.s112y杂合未知错义突变slco1b3外显子5c.c360-3t-纯合良性剪切区变异slco1b3外显子7c.508_509delp.m170fs杂合(疑似)致病移码突变slco1b1外显子8c.g699ap.m233i杂合良性错义突变slco1b3外显子5c.a388gp.n130d杂合良性错义突变serpina1外显子5c.t710cp.v237a杂合良性错义突变
[0110]
患者父亲18c052626的检测结果如表10所示:
[0111]
表10患者父亲18c052626的检测结果
[0112]
突变位置核苷酸改变氨基酸改变纯合/杂合致病性突变类型abcb11外显子22c.2815-8a》g-杂合(疑似)致病剪切区变异abcb11外显子13c.t1331cp.v444a纯合良性错义突变ugt1a1外显子1c.g211ap.g71r杂合未知错义突变ugt1a1外显子4c.c1091tp.p364l杂合未知错义突变slco1b3外显子5c.t334gp.s112a纯合良性错义突变slco1b3外显子5c.c360-3t-纯合良性剪切区变异slco1b3外显子8c.g699ap.m233i纯合良性错义突变slco1b1外显子5c.a388gp.n130d杂合良性错义突变jag1外显子4c.g526ap.v176i杂合疑似良性错义突变
[0113]
患者母亲18c052627的检测结果如表11所示:
[0114]
表11患者母亲18c052627的检测结果
[0115][0116][0117]
三、用于检测或辅助检测遗传性胆红素代谢异常和肝内胆汁淤积症相关基因变异的试剂盒的性能检测
[0118]
1、临床样本测序质量分析
[0119]
按步骤二所述的方法对临床收集的39例全血样本的遗传性胆红素代谢异常和肝内胆汁淤积症相关基因变异进行检测,其中样本编号为g365的测序碱基质量分布图如图1所示,测序深度积累图如图2所示。测序完成后运用trimmomatic进行测序质量分析,其中过滤比率(qcrate)为原始数据过滤后剩余总碱基数(mb)/原始数据总碱基数(mb);深度》=30x的目标区域比率为30x覆盖度,结果如表12。
[0120]
表12测序质量分析结果
[0121]
[0122][0123]
2、临床样本突变位点
[0124]
临床收集的39例临床初诊样本中,gilbert综合症和dubin-johnson综合征患者各13例,bric/pfic i/ii/iii患者7例,alagille综合征和rotor综合征患者各3例,但在39例样本中也检测到除了上述疾病外的其他几个疾病的相关位点,其检测结果如表13所示。
[0125]
表13临床样本基因突变检测结果
[0126]
[0127]
[0128][0129]
39例患者样本共检测到57种突变类型,平均测序深度为548
×
。按市场上测序深度100
×
的要求,使用nextseq
tm
500/550mid output kit v2.5(300cycles)试剂盒一次可以检测超过300例标本。杂合突变频率落在34.3%~61.2%之间,纯合突变频率落在99.6%~100%之间,这完全符合本发明的结果判定要求。
[0130]
由此可见,利用本发明试剂盒进行检测,其准确好、成本低、通量高以及应用于临床研究的可行性好,对临床遗传性胆红素代谢异常和肝内胆汁淤积症的病因起辅助诊断作用。
[0131]
显然,本发明的上述实施例仅仅是为清楚地说明本发明所作的举例,而并非是对本发明的实施方式的限定,对于所属领域的普通技术人员来说,在上述说明的基础上还可以做出其它不同形式的变化或变动,这里无法对所有的实施方式予以穷举,凡是属于本发明的技术方案所引伸出的显而易见的变化或变动仍处于本发明的保护范围之列。
